DNK

Dezoksiribonukleinska kiselina (DNK), nukleinska kiselina koja sadrži uputstva za razvoj i pravilno funkcionisanje svih živih organizama. Zajedno sa Sa RNK i proteinima, DNK je jedan od tri glavna tipa makromolekula koji su esencijalni za sve poznate forme života. Sva živa bića svoj genetički materijal nose u obliku DNK, sa izuzetkom nekih virusa koji imaju ribonukleinsku kiselinu (RNK). DNK ima veoma važnu ulogu ne samo u prenosu genetičkih informacija sa jedne na drugu generaciju, već sadrži i uputstva za građenje neophodnih ćelijskih organela, proteina i RNK molekula. DNK segment koji prenosi ova važna uputstva se naziva gen.[1]
U eukariotima, organizmima kao što su životinje, biljke, gljive i protiste, najveći broj DNK molekula se nalazi u jedru ćelije, a manji broj je u organelama, kao što su mitohondrije ili hloroplasti.[2] U prokariotima (npr. bakterijama) DNK se nalazi u citoplazmi ćelije. Za razliku od enzima, DNK molekul ne utiče direktno na druge molekule, već različiti enzimi sarađuju sa DNK i realizuju informacije bilo u obliku RNK molekula ili u obliku proteina. Ovakav odnos je deo centralne dogme molekularne biologije.[3]
Ćelije sadrže DNK organizovan u duge strukture koje se zovu hromozomi. Tokom pripreme za ćelijsku deobu hromozomi se dupliraju procesom replikacije DNK, tako da svaka od novonastalih ćelija ima kompletan set hromozoma. U hromozomima, hromatinski proteini, kao što su histoni, organizuju DNK na takav način da molekul postaje veoma kompaktan i može da stane u ćelije koje su na hiljade puta manje od raspletenih DNK molekula. Ove kompaktne strukture uslovljavaju interakcije između DNK i drugih proteina, i pomažu u kontrolisanju delova DNK koji se transkribuju.[4]
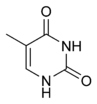
DNK je dugačak polimer, sastavljen od manjih jedinica koje se nazivaju nukleotidi. DNK se sastoji od dva polimerna lanca koji imaju antiparalelnu orijentaciju. Međusobno povezani nukleotidi čine skelet DNK molekula formiran od šećera dezoksiriboze i fosfatnih grupa. Ovaj skelet takođe sadrži četiri različite nukleobaze, vezane za dezoksiribozu. Redosled ove četiri baze je osnova kodiranja genetičkog materijala. Informacija se čita koristeći genetički kod, kojim se specificira sekvenca aminokiselina u proteinima. Kod se čita kopiranjem delova DNK u molekule RNK u procesu koji se naziva transkripcija.
DNK svake osobe, njihov genom, naslijeđen je od oba roditelja. Majčina mitohondrijska DNK zajedno sa 23 kromosoma od svakog roditelja kombinira se u tvorbi genoma zigote tj. oplođene jajne stanice. Kao rezultat, uz pojedine iznimke npr. crvene krvne stanice, većina ljudskih stanica sadrži 23 para kromosoma, zajedno sa mitohondrijskom DNK naslijeđene od majke.
Uvod
DNK nije jedinstvena molekula nego par molekula koje su međusobno povezane vodikovim vezama i organizirane tako da su njeni lanci međusobno komplementarni, od početka do kraja. Svaki se lanac DNK sastoji od građevnih jedinica zvanim nuklotidi kojih ima 4 vrste: adenin (A), citozin (C), guanin (G) i timin (T). U nekim organizmima, posebice u PBS1 faga, uracil zamjenjuje timin u DNK samog organizma. Te osnovne komponente nukleinskih kiselina mogu biti polimerizirane po bilo kojem redu po čemu je sama molekula jedinstvena.
Između dva lanca, svaka baza jednog može biti sparena s određenom bazom drugog lanca i to tako da se adenin spaja uvijek sa timinom (spajaju se sa dvije vodikove veze), i obrnuto te citozin uvijek sa guaninom (tri vodikove veze), i obrnuto. Tako dobivamo moguće kombinacije: A+T, T+A, C+G, G+C. U rijetkim se situacijama događa krivo sparivanje npr. kad timin prijeđe u svoju enolni oblik, a citozin u imino oblik. Dvolančana struktura DNK omogućava jednostavan mehanizam za DNK replikaciju: lanci DNK se odvajaju poput patenta zatvarača čime se lanci otvaraju prema brojnim nukleotidima u okolini. Enzimi stvaraju novi lanac tržeći pravilnu bazu u okolini i sparuju je sa originalnim jednostrukim lancem. Naravno, baza na starom lancu određuje koja će baza biti na novom lancu da se sačuva komplementarnost. Tako stanica završava replikaciju sa još jednom kopijom svoje DNK.
DNK sadrži genetičku informaciju koja se nasljeđuje potomcima. Ta informacija određena je redoslijedom parova baza. Lanac DNK sadrži gene, područja koja reguliraju gene i područja koja nemaju nikakvu funkciju ili funkciju koju mi još ne znamo. Geni se mogu shvatiti kao kuharica ili program organizma.
Još neke odrednice DNK:
- DNK je kiselina zbog fosfatnih skupina između svake deoksiriboze. To je primaran razlog negativnog naboja DNK.
- Polaritet pojedinog para je bitno pa A+T nije isto T+A i C+G nije isto G+C.
- Mutacija su određene greške u lancima DNK koje su nastale slučajno bilo da su baze preskočene, umetnute ili nepravilno udvostručene, zatim lanci mogu biti nadodani ili odrezani ili su to pak kombinacije svih ovih nezgodnih operacija. Mutacije se javljaju pri kemijskim oštećenjima (mutageni), oštećenjima zračenjem (UV zrake) ili kompliciranim zamjenama gena.
- DNK djeluje kao enzim u laboratorijskim uvjetima, ali kod živih organizama to još nije utvrđeno.
- Tradicijski gledano DNK gradi dvostruku formu (Watson-Creekov model), ali može graditi trostruke pa i četverostruke forme (Hoogstenov model).
- DNK se od ribonukleinske kiseline (RNK) razlikuje po tome što sadrži 2-deoksiribozu umjesto riboze te što je timin kod RNK zamijenjen nukleotidom uracilom.
Osobine
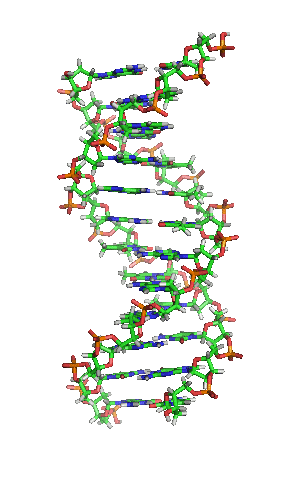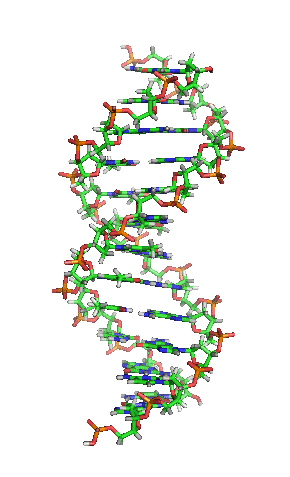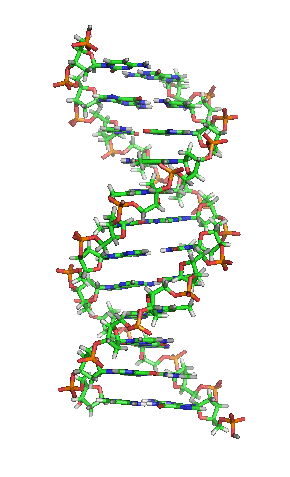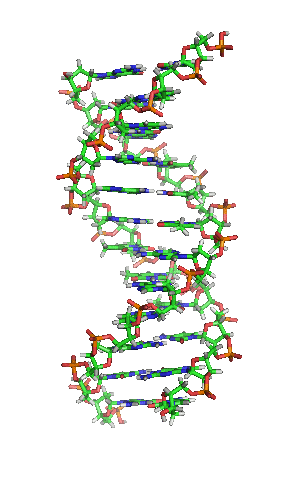

DNK molekul je dugačak polimer koji se sastoji od nukleotida, jedinica koje se ponavljaju.[6][7][8] Nukleotidi su veoma male jedinice, međutim, DNK molekul se sastoji od miliona nukleotida, što ga čini veoma dugim. Najveći ljudski hromozom se sastoji od 440 miliona nukleotida, odnosno 220 miliona parova.[9] Baza koja je povezana sa šećerom naziva se nukleozid, dok baza koja je povezana sa šećerom i jednom ili više fosfatnih grupa se naziva nukleotid.[10] Kada je više nukleotida međusobno povezano, kao npr. u DNK molekulu, taj polimer se onda naziva polinukleotidni lanac.[11]
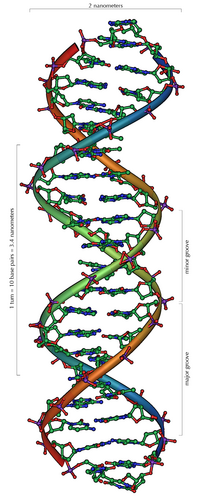
Votson i Krik su 1953. pokazali da je u živim organizmima DNK molekul sastavljen od dva polinukleotidna lanca koji su spiralno uvijeni jedan oko drugog. Za to otkriće su nagrađeni Nobelovom nagradom. Vertikalna dužina svakog obrtaja spirale je 34 angstrema (3,4 nm) i prečnik je 10 angstrema (1,0 nm).[12][13] Prema jednoj drugoj studiji, kada se merenje izvrši u određenom rastvoru DNK lanac je 22 do 26 Angstroma širok (2,2 do 2,6 nm), i jedna nukleotidna jedinica doprinosi dužini sa 3,3 Å (0,33 nm).[14]
Šećer u DNK molekulu je pentoza (nazvan tako jer sadrži pet ugljenikovih atoma) 2-dezoksiriboza (RNK molekul se sastoji od šećera riboze, otuda i pun naziv ribonukleinska kiselina). Šećeri su međusobno povezani fosfatnim grupama koje stvaraju fosfodiestarsku vezu između trećeg i petog ugljenikovog atoma šećernog prstena. Fosfodiestarske veze su asimetrične, te DNK polinukleotidni lanci imaju smer. Kako ovi lanci idu u suprotnim smerovima, kaže se da je DNK antiparalelna. Asimetrični krajevi DNK baza se označavaju sa 5' (pet prim) i 3' (tri prim). Antiparalelnost znači da jedan lanac ide u smeru 5'→ 3', dok suprotni lanac ide u smeru 3'→ 5'. Spiralni lanac koji čini DNK se održava u tom obliku pomoću vodoničnih veza između parova baza.[15] U DNK molekulu postoje četiri baze Adenin (A), Citozin (C), Guanin (G) i Timin (T):[13]
Četiri baze su međusobno komplementarne: adenin (A) jednog lanca je uvek u paru sa timinom (T) naspramnog lanca, i povezani su dvema vodoničnim vezama. Guanin (G) jednog lanca je uvek u paru sa citozinom (C) naspramnog lanca, i povezani su trima vodoničnim vezama. Svaki par baza rotira u odnosu na susedni za 36°, tako da svaki obrtaj spirale dva polinukleotidna lanca čine deset parova baza (A-T i G-C). Polinukleotidni lanci rotiraju u pravcu suprotnom od kazaljki na satu.
Baze su podeljene u dve grupe – purinske (adenin i guanin) i pirimidinske (citozin i timin). Uracil (U) baza koja se nalazi u RNK molekulu i koja zamenjuje timin, pripada pirimidinskoj grupi. Jedina razlika između timina i uracila je nedostatak jedne metil-grupe kod uracila. Uracil je standardna baza RNK molekula, i ne postoji kao takva u DNK molekulu. Jedini, trenutno poznati, izuzetak je bakterijski virus PBS1 koji u svom DNK molekulu ima uracil kao sastavnu bazu[16]. Osim RNK i DNK veliki broj veštačkih analoga nukleinskih kiseline je kreiran radi studiranja osobina nukleinskih kiselina, kao i za primenu u biotehnologiji.[17]
Žlebovi

Dva komplementarna heliksna lanca formiraju osnovu DNK molekula. Prepoznatljiv je i dvostruki heliks koji prati otvore, ili žlebove, između lanaca. Ta udubljenja su neposredno pored parova baza, te mogu da služe kao mesta vezivanja proteina i malih molekula. Pošto lanci nisu direktno nasuprot jedan drugom, žlebovi nemaju jednaku veličinu. Veliki žleb je 22 Å širok, dok mali žleb ima 12 Å.[21] Postoji niz izuzetaka, prvenstveno u slučajevima neobičnih DNK konformacija. Nazivi veliki i mali žleb se uvek odnose na razlike u veličini udubljenja kad je DNK u običnoj B formi.
Baze malog žleba su na podesnijem rastojanju za vezivanje liganda, nego baze velikog žleba. Iz tog razloga, proteini poput traskripcionih faktora koji se vezuju za specifične sekvence dvostrukog DNK heliksa obično formiraju vodonične veze sa izloženim stranama baza malog žleba.[22]
Denaturacija i hibridizacija DNK
Sekundarna struktura DNK je podložna denaturaciji. Pod denaturacijom se podrazumeva narušavanje sekundarne strukture tako da se dvolančani DNK molekul razdvaja na dva polinukleotidna lanca. Pod odgovarajućim uslovima može doći do renaturacije, tj. do ponovnog spajanja komplementarnih lanaca DNK.[23] Procesi denaturacije i renaturacije odigravaju se i u ćeliji pod kontrolisanim uslovima i u ograničenom obimu. Ti procesi predstavljaju neophodan preduslov za normalno funkcionisanje DNK, odnosno za njenu replikaciju i transkripciju.
Kada se u rastvoru nađu dva polinukleotidna lanca koji imaju komplementarne redoslede nukleotida, nagradiće se hibridni dvolančani molekul.[24][25] Denaturisana DNK može da hibridizuje sa denaturisanom DNK iste ili različite vrste, ili sa RNK. Hibridizacija je našla veoma široku primenu u istraživanjima u molekularnoj biologiji i predstavlja jednu od osnovnih tehnika genetičkog inženjeringa.
Sparivanje baza


|

|
Svaka baza jednog lanca se sparuje samo sa jednom bazom na naspramnom lancu. Ovakvo sparivanje se naziva komplementarno sparivanje. Purin se sparuje sa pirimidinom vodoničnim vezama, te se A sparuje samo sa T (dvema vodoničnim vezama) i C samo sa G (sa tri vodonične veze). Kako vodonične veze nisu kovalentne, one se lako raskidaju i lako ponovo formiraju. Ove veze se raskidaju ili mehaničkim silama (npr. tokom replikacije) ili visokom temperaturom.[26] Dve vodonične veze se lakše raskidaju od tri. Ovaj podatak je bitan ako sekvenca DNK molekula nije unapred poznata. Kad je sekvenca DNK molekula nepoznata, u molekularnoj biologiji se između ostalog primenjuje tehnika koja koristi temperaturu. Što je temperatura viša, to se DNK molekul teže raskida, te se može pretpostavi da taj molekul DNK ima veliki broj C i G baza (i.e. visok GC-sadržaj). DNK sa visokim GC-sadržajem je stabilnija od DNK sa niskim GC-sadržajem.[27]
Direktna posledica nukleotidne komplementarnosti je da su informacije u dvolančanoj sekvenci DNK heliksa duplirane, što je vitalno za replikaciju molekula. Ova reverzibilna i specifična interakcija između komplementarnih parova baza je kritična za sve funkcije DNK u živim organizmima.[7]
Kao što je gore napomenuto, većina DNK molekula se sastoji od dva polimerna lanca, spojena u heliksnu strukturu nekovalentnim vezama. Ova dvolančana struktura (dsDNA) se u znatnoj meri održava posredstvom interakcija međulančanog slaganja baza, koje se najjače za G,C stekove. Dva lanca se mogu razdvojiti u procesu poznatom kao topljenje, čime se formiraju dva molekula (ssDNA). Do topljenja dolazi kad su uslovi podesni, kao što su visoke temperature, niske koncentracije soli i visoke pH vrednosti (nizak pH takođe otapa DNK, ali pošto je DNK nestabilna usled denaturacije kiseline, nizak pH se retko koristi).
Stabilnost dsDNA forme zavisi ne samo od GC-sadržaja (% G,C baznih parova) nego i od sekvence (pošto je formiranje stekova zavisno od nje), kao i od dužine (duži molekuli su stabilniji). Stabilnost se može meriti na različite načine. Uobičajen pristup je merenje temperature topljenja (ona se još naziva Tm vrednost), što je temperatura na kojoj se 50% dvolančanih molekula konvertuje u jednolančane molekule. Nastali jednolančani DNK molekuli nemaju jedinstven zajednički oblik, mada su neke konformacije stabilnije od drugih.[28] Temperatura topljenja je zavisna od jonske snage i DNK koncentracije. Konsekventno, GC sadržaj i dužina dvostrukog DNK lanca određuju jačinu asocijacije između lanaca. Dugi DNK heliksi sa visokim GC sadržajem imaju lance sa jačim interakcijama, dok kratki heliksi sa visokim AT sadržajem imaju lance sa slabijim interakcijama.[29] U biologiji, delovi DNK dvostrukih heliksa koji se lako razdvajaju, kao što je TATAAT Pribnov-kutija u nekim promoterima, teže da imaju visok AT sadržaj.[30][31][32]
Smisao i antismisao
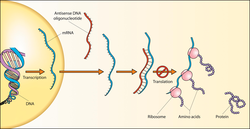
DNK sekvenca se naziva smisaonom (engl. sense) (negativna (-)), ako je njena sekvenca ista kao i sekvenca iRNK kopije koja se translira u protein.[33] Komplementarna sekvenca suprotnog lanca se naziva antismisaona (pozitivna (+)') sekvenca. Obe sekvence mogu da postoje na različitim delovima istog DNK lanca (i.e. oba lanca mogu da sadrže obe smisaone i antismisaone sekvence). Ponekad se fraza kodirajući lanac sreće. Međutim, kodirajuća i nekodirajuća RNK mogu da budu transkribovane na sličan način sa oba lanca. U nekim slučajevima do transkripcije dolazi u oba pravca počevši od zajedničkog promoterskog regiona, ili do transkripcije može doći unutar introna, na oba lanca.[34][35][36]
Kod prokariota i eukariota, antismisaone RNK sekvence se formiraju, mada funkcija tih molekula nije potpuno jasna.[37] Jedna od pretpostavki je da antismisaoni RNK molekuli učestvuju u regulaciji ekspresije gena putem RNK-RNK sparivanja baza.[38]
Kod malog broja DNK sekvenci prokariota u eukariota, i nešto većeg broja plazmida i virusa, razlika između smisaonih i antismisaonih lanaca je zamagljena preklapanjem gena.[39] U tim slučajevima, neke DNK sekvence imaju dvostruku ulogu. One kodiraju jedan protein kad se čitaju duž jednog lanca, i drugi protein kad se čitaju u suprotnom pravcu duž drugog lanca. Kod bakterija, ovo preklapanje može da učestvuje u regulaciji transkripcije gena,[40] dok kod virusa, preklapajući geni povećavaju količinu informacije koja može da bude kodirana unutar malog viralnog genoma.[41]
Pakovanje DNK molekula u ćelijama
| Nukleozom | |||||
|---|---|---|---|---|---|

| |||||
| Kristalna struktura nukleozoma. Proteinske komponente su H2A , H2B , H3 i H4 . Pogled je odozgo kroz superheliksnu osu. (1KX5)[42] | |||||
| Identifikatori | |||||
| Simbol | Histone | ||||
| Pfam | PF00125 | ||||
| Pfam klan | CL0012 | ||||
| InterPro | IPR007125 | ||||
| SKOP | 1hio | ||||
| |||||
Gotovo kod svih prokariota, DNK je kružni molekul sagrađen od dva spiralno uvijena polinukleotidna lanca. Kod eukariota organizacija DNK molekula je nešto komplikovanija. DNK molekul je veoma dugačak, u proseku do 1,8 metara. Molekul te dužine mora da stane u ćelije koje su veoma male i ne mogu da se vide golim okom. Ćelije moraju da veoma kompaktno spakuju DNK molekul. To omogućavaju proteinima koji se zovu histoni.[43][44]
Histoni su mali, veoma bazni proteini, bogati amino kiselinama kao što su lizin i arginin. Oni su glavne proteinske komponente hromatina, koje deluju kao kalemi oko kojih se namotava DNK. Oni učestvuju u regulaciji genske aktivnosti. Bez histona, nesavijena DNK u hromozomima bi bila veoma dugačka (odnos dužine i širine je veći od 10 miliona kod ljudske DNK). DNK namotana na histone proizvodi oko 90 mikrometera (0.09 mm) hromatina, koji se duplira i kondenzuje tokom mitoze, dajući oko 120 mikrometara hromozoma.[45]
U eukariotskim ćelijama je postoji pet tipova histona: H1, H2A, H2B, H3 i H4.[46][47] Histoni su u direktnom kontaktu sa DNK molekulom. Osam histona (po dva H2A, H2B, H3, H4), stvaraju strukture koje izgledaju kao disk. Struktura DNK molekula obavijenog oko diska se naziva nukleozom. Oko svakog diska DNK molekul se obavije 1,65 puta, u dužini od 147 baznih parova (A-T i C-G), formirajući levoruki superheliksni namotaj.[48] Tako uvijeni DNK molekul se obavije oko preostalog histona H1, koji ne formira strukturu u obliku diska, već služi samo kao veza do sledećeg diska (izgrađenog od gorepomenutih histona) i ponovo se obavija oko sledećeg diska.[49] Histon H1 omogućava formiranje strukture višeg reda. Najosnovnija takva formacija je vlakno prečnika 10 nm. Gledano kroz mikroskop sveukupna ovakva struktura izgleda kao perlana ogrlica. Ovo uključuje pakovanje DNK oko nukleozoma sa oko 50 baznih parova između njih (ti segment se nazivaju linker DNK).
Osnova nukleozoma je formirana od dva H2A-H2B dimera i H3-H4 tetramera. Oni formiraju dve skoro simetrične polovine tercijarne strukture, sa C2 simetrijom, gde je jedan makromolekul slika u ogledalu drugog molekula.[48] Četiri osnovna histona (H2A, H2B, H3 i H4) imaju relativno slične strukture i visoko su očuvani tokom evolucije. Svi imaju heliks obrt heliks obrt heliks motiv (koji omogućava laku dimerizaciju). Oni isto tako imaju duge repove na jednom od krajeva aminokiselinskog lanca, na kojima dolazi do niza posttranslacionih modifikacija.
Smatra se da su histonski proteini evoluciono srodni sa heliksnim delom produženog AAA+ ATPaznog domena,[50][51] C-domena, i sa N-terminusnim domenom prepoznavanja supstrata Clp/Hsp100 proteina.[52][53] Uprkos razlikama u njihovoj topologiji, oni imaju homologan heliks-ravan-heliks (HSH) motiv.[54]
Koristeći tehniku spin-obeležene elektronske paramagnetne rezonance, Britanski istraživači su izmerili rastojanje između namotaja oko kojih eukariotske ćelije namotavaju svoju DNK. Utvrđeno je da su rastojanja u opsegu od 59 do 70 Å.[55]
Histoni formiraju pet tipova interakcija sa molekulom DNK:
Visoko bazna priroda histona, osim što omogućava DNK–histon interakcije, doprinosi njihovoj rastvorljivosti u vodi.
Superspiralizacija


DNK može da bude uvijena poput kanapa procesom koji se naziva superspiralizacija DNK. Kad je DNK u svom „opuštenom“ stanju, lanci obično obiđu osu dvostrukog heliksa jednom svaka 10,4 bazna para, dok ako je DNK upredena lanci postaju više ili manje zbijeni.[56] Kada je DNK upredena u pravcu heliksa, u pitanju je pozitivna superspiralizacija i baze su bliže jedna drugoj. Ako je DNK upredena u suprotnom pravcu (negativna superspiralizacija), baze se lakše rastavljaju. U prirodi, DNK je najčešće blago negativno superspiralizovana. To se ostvaruje enzimima koji se zovu topoizomeraze.[57] Ti enzimi su isto tako potrebni za otpuštanje naprezanja usled DNK uvijanja nastalog tokom procesa transkripcije i replikacije DNK.[58][59]
Hromozomi mogu da budu veoma veliki, te središnji segmenti mogu da se ponašaju kao da su krajevi učvršćeni. Rezultat toga je da oni ne mogu da raspodele suvišne namotaje na ostatak hromozoma, ili da apsorbuju zavijanje da bi se oporavili od odvijanja, te segmenti mogu da postanu superspiralizovani. U odgovoru na superspiralizaciju oni će biti izloženi naprezanju, kao da su krajevi spojeni.
Superspiralizacija DNK je važna za njeno pakovanje unutar ćelija. Dužina nespiralizovane DNK je hiljadama puta veća od dužine ćelije, te je pakovanje genetičkog materijala unutar ćelije ili jedra (kod eukariota) kompleksan zadatak. Superspiralizacija DNK redukuje potrebni prostor i omogućava pakovanje znatno veće količine DNK. Kod prokariota, plektonemski supernamotaji su predominantni, zato što je hromozom najčešće kružnog oblika i sadrži relativno malu količinu genetičkog materijala.[60][61][62][63][64] Superspiralizacija DNK kod eukariota se javlja na više nivoa plektonemskih i solenoidnih supernamotaja, pri čemu se solenoidna superspiralizacija pokazuje najefektivnijom u zbijanju DNK molekula. Solenoidna superspiralizacija se ostvaruje putem histona i formira se vlakno prečnika 10 nm. Ovo vlakno se dalje namotava u 30 nm široko vlakno, koje se dalje namotava na samo sebe više puta.[65][66][67]
DNK pakovanje se odvija u znatno povećanoj meri tokom deobe jedra u procesima mitoze ili mejoze, pri kojim se DNK mora podeliti i sažeti u novonastalim ćelijama. Kondenzini[68][69] i kohezini[70][71][72] su proteini za strukturno održavanje hromozoma koji pomažu u kondenzaciji hromatida i vezivanju njihovih centromera. Ti proteini indukuju pozitivnu superspiralizaciju.[73][74][75]
Alternativne DNK strukture



DNK može da postoji u mnoštvu mogućih konformacija među kojima su A-DNK,[76] B-DNK,[77][78][79] i Z-DNK forme[80][81], mada su jedino B-DNK i Z-DNK direktno primećene u funkcionalnim organizmima.[10] Konformacija koju DNK poprima zavisi od nivoa hidratacije, DNK sekvence, količine i pravca supernamotavanja, hemijskih modifikacija baza, tipa i koncentracije metalnih jona, kao i prisustva poliamina u rastvoru.[82]
Prvi objavljeni izveštaji o Rendgenskoj strukturi A-DNK i B-DNK formi su koristili analizu baziranu na Patersonovoj transformaciji koja daje samo ograničenu količinu strukturne informacije o orijentaciji DNK vlakana.[83][84][85] Jedan alternativni analitički pristup su predložili Vilkins et al. 1953, za in vivo B-DNK difrakciju X-zraka/rasporeda maksimuma rasejavanja visoko hidratisanih DNK vlakana baziran na kvadratima Baselovih funkcija.[86] U istom žurnalu, Džejms D. Votson i Fransis Krik su objavili njihovu analizu molekulskog modelovanja DNK difrakcionih obrazaca X-zraka i predložili strukturu dvostrukog heliksa.[12]
Mada je B-DNK forma najčešća pod uslovima koji vladaju u ćelijama,[87] ona nije dobro definisana konformacija nego je familija srodnih DNK konformacija[88] koja se javlja pri visokim nivoima hidratacije prisutnim u živim ćelijama. Njihovi odgovarajući rendgenski difrakcioni obrasci rasipanja su karakteristični za molekulske parakristale sa znatnim stepenom nereda.[89][90]
U poređenju sa B-DNK, A-DNK forma je šira desnoruka spirala, sa plitkim, širokim glavnim žlebom i užim, dubljim malim žlebom. A forma se javlja pod nefiziološkim uslovima u parcijalno dehidratisanim DNK uzorcima, dok se u ćeliji može formirati pri hibridnom sparivanju DNK i RNK lanaca,[91][92][93] kao i u enzim-DNK kompleksima.[94][95] Segmenti DNK gde su baze hemijski modifikovane metilacijom mogu da podlegnu većim konformacionim promenama i da poprime Z formu. Ovde, lanci formiraju levoruku spiralu oko heliksne ose, što je suprotno uobičajenoj B formi.[96] Te neobične strukture se mogu prepoznati po specifičnim Z-DNK vezujućim proteinima. One mogu da učestvuju u regulaciji transkripcije.[97][98][99][100][101][102]
Alternativna DNK hemija

Tokom dužeg niza godina egzobiolozi su predlagali postojanje jedne alternativne biosfere koja koristi radikalno različite biohemijske i molekulske procese nego trenutno poznate životne forme. Jedna od pretpostavki je bila postojanje životnog oblika koji koristi arsenik umesto fosfora u DNK.
Na jednoj konferenciji za štampu NASA je decembra 2010. izjavila da bakterija GFAJ-1, koja je evoluirala u okruženju bogatom u arseniku, prva zemaljska životna forma koja možda ima tu sposobnost.[103] Bakterija je nađena u Mono jezeru, istočno od Jesemitskog nacionalnog parka. GFAJ-1 je štapićasta ekstremofilna bakterija iz familije Halomonadaceae, koja u odsustvu fosfora možda ima sposobnost inkorporisanja obično otrovnog elementa arsena u svoj DNK.[104] Ovo otkriće ide u prilog dugogodišnjoj ideji da bi vanzemaljski život možda mogao da ima različitu biohemijsku osnovu od života na Zemlji.[104][105] Istraživanja je izveo tip predvođen Felisom Volf-Simon, koja je geomikrobiolog i geobiohemičar na NASA astrobiološkom institutu pri Državnom univerzitetu Arizone.
Ovaj nalaz je naišao na jak kriticizam u naučnoj zajednici. Naučnici tvrde da nema dokaza da je arsenik zapravo inkorporiran u biomolekule.[105][106] Mikrobiolog Johan Hajder je kritikovao prezentovane rezultate studije. On je uputio na moguće greške u merenju kao i na pogrešnu interpretaciju rezultata studije. Po njemu je, u originalnoj publikaciji autora pomenuto zagađenje uzoraka ostacima fosfata, koji su verovatno prisutni u dovoljnoj količini za osnovno snabdevanje bakterija.[107] Nezavisno potvrđivanje ovog do sad nije bilo moguće.
Kvadrupleksne strukture




Na krajevima linearnih hromozoma su specijalizovani regioni DNK koji se nazivaju telomere.[109][110][111] Glavna funkcija tih regiona je da se omogući ćeliji da replikuje krajeve hromozoma koristeći enzim telomerazu, pošto enzimi koji normalno replikuju DNK ne mogu da kopiraju ekstremne 3′ krajeve hromozoma.[112][113] Ti specijalizovani hromozomski završeci takođe pomažu u zaštiti DNK krajeva, i sprečavaju sisteme za popravku DNK u ćeliji da ih tretiraju kao oštećenja koja treba popravljati.[114][115][116] U ljudskim ćelijama, telomere su obično segmenti jednolančane DNK koji se sastoje od nekoliko hiljada ponavljanja jednostavne TTAGGG sekvence.[117][118][119]
Ove guaninom bogate sekvence mogu da stabilizuju hromozomske krajeve formiranjem struktura svežnjeva jedinica sa četiri baze, umesto uobičajenih baznih parova DNK molekula. Ovde, četiri guaninske baze formiraju ravnu površinu, i te ravne četvorobazne jedinice se zatim slažu jedna na drugu da formiraju stabilne G-kvadrupleksne strukture (G-tetrade G4-DNK).[120][121] Ove strukture su stabilizovane vodoničnim vezivanjem između baza i helacijom metalnog jona u centru svake četvorobazne jedinice.[122] Niz drugih strukture se može formirati, sa centralnim setom od četiri baze koje dolaze bilo iz jednog lanca savijenog oko baza, ili nekoliko različitih paralelnih lanaca, pri čemu svaki doprinosi jednu bazu centralnoj strukturi.
Osim ovih stekovanih struktura, telomere isto tako formiraju strukture sa velikim petljama koje se nazivaju telomerne petlje, ili T-petlje. Ovde se jednolančana DNK sklupča u veći krug stabilizovanom proteinima koji se vezuju za telomere.[123] Na samom kraju T-petlje, jednolančana telomerna DNK je spojena sa regionom dvolančane DNK tako što telomerni lanac delom remeti strukturu DNK dvostrukog heliksa i bazno se sparuje sa jednim od dva lanca. Ova trolančana struktura se naziva deplasmanska petlja ili D-petlja.[120]
Kvadrupleksi se javljaju ne samo u telomerama, nego i na drugim lokacijama. Na primer, za protoonkogen c-myc je pokazano[124][125] da formira kvadrupleks u nukleaznom hipersenzitivnom regionu[126][127], koji je kritičan za aktivnost gena.[128] Nakon tog inicijalnog otkrića, za mnoge druge gene je nađeno da imaju G-kvadruplekse u njihovim promoterskim regionima.[129] Neki od njih su živinski β-globinski gen,[130][131][132] ljudska ubikvitinska ligaza RFP2[133][134][135] i protoonkogeni c-kit,[136][137][138] bcl-2[139][140][141], VEGF,[142][143][144] H-ras[145][146][147] i N-ras.[148][149][150]
Identifikacija i predviđanje sekvenci koje imaju sposobnost formiranja kvadrupleksa je važan korak u razumevanju njihove uloge. Generalno jednostavni obrasci se koriste za pretragu mogućih kvadrupleks formirajućih sekvenci: d(G3+N1-7G3+N1-7G3+N1-7G3+), pri čemu je N baza (uključujući guanin).[151][152] Ovo pravilo je našlo široku primenu u onlajn algoritmima.
Pregledi celokupnog genoma bazirani na pravilu nalaženja kvadrupleksa su identifikovali 376.000 mogućih kvadrupleksnih sekvenci (PQS) u ljudskom genomu. Znatan broj njih se verovatno ne formira in vivo.[152] Jedna slična studija je identifikovala moguće G-kvadrupleks kod prokariota.[153] Postoji više modela koji objašnjavaju kako kvadrupleksi mogu da kontrolišu aktivnost gena. Jedan model pokazuje da formiranje G-kvadrupleksa na ili blizo promotera blokira transkripciju gena, i time ga deaktivira.[154][155][156] U jednom drugom modelu kvadrupleks formiran na nekodirajućem DNK lancu pomaže u održavanju otvorene konformacije kodirajućeg DNK lanca, te pospešuje ekspresiju respektivnog gena.[157][158][159]
Jedan način indukovanja ili stabilizovanja G-kvadrupleksnih formacija, je uvođenje molekula koji se mogu vezati za G-kvadrupleksne strukture.[160][161] Poznati su brojni ligandi, mali molekuli i proteini koji imaju tu sposobnost. Znatan broj proteina koji se javlja u prirodi se selektivno vezuje za G-kvadruplekse. Među njima su helikaze,[162][163][164] koje su implicirane u Blumov[165][166] i Vernerov sindrom[165][167], i protein RAP1 iz Saccharomyces cerevisiae.[168][169][170] Razvijen je protein sa domenom cinkovog prsta[171] koji se nazva Gq1,[172][173][174] kao i specifična antitela.[175][176][177]
Poznato je da se katjonski porfirini vezuju za G-kvadruplekse,[178][179][180] kao i molekul telomestatin.[181][182][183]
Razgranata DNK

|

|
| Jedna grana | Višestruko grananje |
Do DNK krzanja dolazi kad nekomplementarni regioni postoje na jednom ili oba kraja inače komplementarne dvolančane DNK. Razgranata DNK se može javiti ako se uvede treći DNK lanac koji ima sposobnost hibridizacije sa otvorenim DNK segmentima dvolančane DNK. Najjednostavniji primer razgranavanja je trolančana DNK. Kompleksi sa dodatnim lancima i višestrukim grananjem su takođe poznati.[185] Razgranata DNK nalazi primenu u nanotehnologiji.
Test razgranate DNK je test amplifikacije signala (za razliku od testa amplifikacije biološke mete) koji se koristi za detektovanje molekula nukleinskih kiselina.[186] Ovaj test se može koristiti za detektovanje i kvantifikaciju mnogih tipova RNK ili DNK. U testu se razgranata DNK pomeša sa testiranim uzorkom. Detekcija se vrši koristeći neradioaktivni metod. Prethodna amplifikacija nukleinske kiseline nije neophodna. Test je u potpunosti zavistan od hibridizacije. Enzimi se koriste za određivanje stepena hibridizacije, ali se ne koriste za manipulaciju nukleinskih kiselina. Male količine nukleinske kiseline se mogu detektovati i kvantifikovati bez koraka reverzne transkripcije (u slučaju RNK) i/ili PCR.[187][188] Test je podesan za upotrebu u visoko protočnom modu, za razliku od kvantitativnog Northern-blota[7][189] ili testa RNK protekcije.[190]
Vibracije
DNK može da izvodi nisko frekventno kolektivno kretanje. Ono se može meriti Ramanovom spektroskopijom[191][192] i analizirati primenom modela kvazi kontinuuma.[193][194]
Kružna DNK
Kad su krajevi komada dvostruke zavojnice DNK spojeni tako da tvore krug, kao u plazmida, lanci su topološki zamršeni. To znači da ne mogu biti razdvojeni laganim zagrijavanjem ili bilo kojim procesom koji ne uključuje prekidanje lanaca. Enzimi topoizomeraze su zaslužni za odmrsavanje topološki povezanih lanaca. Neki enzimi to čine cijepanjem dvaju lanaca tako da i drugi dvolančani segment može proći. Odmrsavanje je potrebno za replikaciju kružne DNK kao i za različite tipove linearnih DNK.
Entropijsko rastezanje
Kada se DNK nalazi u otopini, podvrgnuta je komformacijskim kolebanjima zbog energije koja se nalazi u samoj otopini. Zbog entropijskih razloga, savitljiva stanja su termički pogodnija od rastegnutih stanja. Zato se DNK rasteže slično gumenoj traci. Koristeći optička kliješta, entropijsko rastezanje DNK je analizirano iz perspektive fizike polimera i utvrđeno je da se DNK ponaša kao Kratky-Porodov crvoliki lanac, model sa duljinom postojanosti od oko 53 nm.
DNK se zatim podvrgava rastezanju faznog prijelaza pri sili od 65 pN. Pri višim vrijednostima sile, DNK poprima oblik koji je pretpostavio Linus Pauling tako da se fosfati nalaze u sredini, a baze su okrenute prema van. Ta predložena struktura se naziva P-oblik DNK u čast Paulingu.
Superzavojnica
B oblik DNK zavojnice se zakreće 360° po 10 bp u odsutnosti naprezanja. Ali mnogi molekularni biološki procesi mogu izazvati to naprezanje. To će rezutirati prevelikim ili premalim zavojima, odnosno kao pozitivno ili negativno superzavijanje. DNK je in vivo tipično negativno superzavijena, što ubrzava odmotavanje dvostrukog heliksa za transkripciju.
Nabor šećera
Postoje 4 konformacije prstena ribofuranoze u nukleotidu:
- C-2' endo
- C-2' egzo
- C-3' endo
- C-3' egzo
Riboza je inače u C-3’ endo, dok su deoksiriboze inače u C-2’endo konformaciji šećernog nabora. A i B oblici se uglavnom razlikuju po njihovim šećernim oblicima. U A obliku, C3’ konfiguracija je iznad prstena šećera dok je kod C2’ konfiguracije ispod. Tako se A oblik opisuje kao C-3’ endo. Isto tako, u B obliku, C2’ konfiguracija je iznad prstena šećera, a C3’ ispod pa se naziva C-2’ endo. Drukčije nabiranje A-DNK rezultira skraćenju udaljenosti između susjednih fosfata za 1 angstrem. To daje 11 ili 12 parova baza u zavoju DNK lanca, umjesto 10.5 u B-DNK. Šećerni nabor daje DNK jednoliki oblik vrpce, sa cilindrično otvorenim središtem i skučenijim, izraženim dubljim glavnim utorom nego što su utori u B-DNK.
A i Z oblici zavojnice
Dva ostala poznata geometrijska oblika (A i Z) razlikuju se u njihovoj geometriji i dimenzijama. A oblik se nalazi samo u dehidriranim uzorcima DNK, kao što su oni koji se nalaze u kristalografskim eksperimentima te u hibridno sparenih DNK i RNK lanaca. Segmenti DNK koje je stanica metilirala u regulacijske svrhe pripadaju Z geometriskom obliku u kojem se lanci okreću oko osi zavonice kao zrcalna slika B oblika.
Odlike različitih oblika zavojnica
| Geometrijska osobina | A-forma | B-forma | Z-forma |
|---|---|---|---|
| Smjer zavojnice | desni vijak | desni vijak | lijevi vijak |
| Jedinica ponavljanja | 1 bp | 1 bp | 2 bp |
| Rotacija/bp | 33.6° | 35.9° | 60°/2 |
| Broj parova baza po zavoju | 10.7 | 10.4 | 12 |
| Inklinacija bp od osi | +19° | -1.2° | -9° |
| Rast/bp duž osi | 0.23 nm | 0.332 nm | 0.38 nm |
| Period po okretu zavojnice | 2.46 nm | 3.32 nm | 4.56 nm |
| Srednji okret vijka | +18° | +16° | 0° |
| Glikozilni kut | anti | anti | C: anti, G: syn |
| Šećerni nabor | C3'-endo | C2'-endo | C: C2'-endo, G: C2'-exo |
| Promjer | 2.6 nm | 2.0 nm | 1.8 nm |
Treba spomenuti da postoje i neki nezavojiti oblici DNK npr. SBS (side-by-side; usporedna) konfiguracija DNK.
Uloga redoslijeda
Unutar gena, redoslijed nukleotida duž DNK određuje glasničku RNK (eng. messenger RNA) koja pak definira protein kojeg je organizam dužan obraditi izraziti na određena mjesta tijekom života. Translacija proces kojim se odvija sinteza proteina pomoću redoslijeda aminokiselina određene redoslijedom nukleotida. Taj redoslijed nukleotida naziva se genetički kod, a sastoji se od tri nukleotida i naziva se kodon kojeg opisujemo trima slovima imena baza (npr. ACT, CAG, TTT). Ti kodoni traslacijom stvaraju glasničku RNK (mRNK) i tada transportna RNK (eng. transfer RNA), tRNK, prema odgovarajućem kodonu dodaje odgovarajuću aminokiselinu. Moguće je 64 kodona (4 vrste baza za tri mjesta kodona: 43) koji kodiraju 20 aminokiselina. Više kodona može kodirati jednu te istu aminokiselinu, a postoje i ‘’stop’’ ili nonsensni kodoni koji označavaju kraj kodne regije (UAA, UGA i UAG kodoni).
U mnogih vrsta, samo mali dio ukupnog redoslijeda genoma kodira protein. Na primjer, samo 1.5% humanog genoma se sastoji od dijelova koji kodiraju proteine, a to su egzoni. Funkcija ostalih dijelova je manje poznata. Postoje redoslijedi DNK koji imaju specifični afinitet za proteine koji vežu DNK (eng. DNA binding proteins) koji igraju veliku ulogu u replikaciji i tanskripciji. Takvi redoslijedi DNK nazivaju se regulacijske sekvence i istraživači pretpostavljaju da su našli tek mali dio takvih sekvenci od ukupnog broja. Otpadna DNK (eng. junk DNA) predstavlja redoslijede u kojima nema gena i nemaju nikakvu funkciju. Razlozi postojanja toliko mnogo nekodirajuće DNK u eukariotskim genomima i iznimne razlike veličine genoma (C – veličina) između vrsta predstavljaju problem nazvan enigma C-veličine.
Neke sekvence DNK igraju strukturnu ulogu u kromosomima. Telomere i centromere sadrže malo ili uopće ne sadrže gene za kodiranje proteina, ali su važne za funkciju i stabilnost kromosoma. Neki RNK geni kodiraju traskripte koji funkcioniraju kao regulatorne DNK koje utječu na funkciju drugih DNK molekula. Intrinska i egzonska struktura nekih gena (geni imunoglobulina i protokadeherina) su važni za dopuštanje alternativnog izrezivanja (splicing) pre-mRNK te se pri tome stvaraju različiti proteini koji u biti potječu od jednog gena. Neke nekodirajuće regije predstavljaju pseudogene koji mogu biti korišteni kao materijal za stvaranje novih gena s novim funkcijama. Postoje i nekodirajuće regije koje omogućuju vruće točke za duplikaciju kratkih dijelova DNK te takve duplicirane sekvence mogu biti glavni oblik genetičke promijene u ljudskom porijeklu. Egzoni među kojima je mnoštvo introna omogućuju ‘’egzonsku prevrtljivost’’ pri stvaranju modificiranih gena koji mogu imati novu prilagodbenu funkciju. Velika količina nekodirajuće DNK su vjerojatno prilagodbeni tako što omogućuju kromosomskim regijama gdje se vrši rekombinacija između homolognih dijelova kromosoma bez poremećaja u funkciji gena. Redoslijedi DNK također određuju podložljivost cijepanju restrikcijskim enzimima vrlo bitnim za genetički inženjering. Točno mjesto cijepanja nečijeg genoma vrstu ‘’DNK otiska’’ pojedine individue.
Smjer lanaca

Asimetrični oblik i povezanost nukleotida znači da DNK lanac uvijek ima određenu orijentaciju i usmjerenost. Zbog usmjerenosti, blizak uvid u dvostruku zavojnicu otkriva da nukleotidi jednoga lanca prate jedan put (acendentni lanac) odnosno lanac ‘’raste’’, a nukleotidi drugog lanca drugi put (descendentni lanac) odnosno taj lanac ‘’opada’’. Tako izgleda da su lanci antiparalelni.
Hemijska nomenklatura (5’ i 3’ krajevi)
U svakoj DNK postoje asimetrični krajevi pri čemu kraj prvog lanca naziva 5’ kraj, a kraj drugog lanca 3’ kraj. Unutar stanice, enzimi koji izvode replikaciju i transkripciju čitaju DNK uvijek od 3’ prema 5’ smjeru jednog lanca, dok enzimi koji provode translaciju (na RNK) čitaju u suprotnom smjeru. U laboratorijskim uvjetima moguće su i manipulacije smjera čitanja. U vertikalno orijentiranoj dvostrukoj zavonici kažemo da lanac od 3’ kraja raste, a drugi lanac od 5’ kraja opada.
"Sense" i "antisense"
Rezultat antiparalelnog ustroja lanaca i odlika enzima koji čitaju sekvence DNK je taj da stanice mogu pravilno translatirati samo jedan od njih. Drugi se lanac može čitati samo unatrag. Prema molekularnim biolozima sekvenca je smislena (‘’sense’’) ako može biti prevedena, a njezina komplementarna sekvenca je nelogična/besmislena (‘’antisense’’). Prema svemu ovome podloga za transkripciju je smislena sekvenca, a transkript smislenog lanca je i sam po sebi smislen.
Razlike među sense i antisense sekvenci
U malog udijela gena prokariota, te više u virusa i plazmida postoje male razlike između sense i antisense lanaca. Određene sekvence njihovog genoma ima dvostruku zadaću da očitavaju jedan lanac u smjeru 5’ prema 3’ te drugi lanacu smjeru 3’ prema 5’. Kao rezultat toga, genomi tih virusa su neuobičajeno kompaktni za brojne gene koje sadrže, za koje biolozi vjeruju da predstavljaju prilagodbu. To jednostavno potvrđuje da nema biološlike među dvama lanaca dvostruke zavojnice. Tipično je da se svaki lanac DNK ponaša kao sense i antisense u različitim regijama.
Jednolančana DNK, ssDNK (eng. single stranded DNA)
U nekim se virusima DNK javlja u nezavojitom jednolančanom obliku. Zbog mnogih mehanizama popravka DNK u stanici koji djeluju samo na uparenim bazama, u virusa koji nose jednolančanu DNK genomi mutiraju učestaluje. Takve se vrste mnogo brže prilagođavaju i odupiru izumiranju. Rezultat ne bi bio zadovoljavajući u kompliciranijih i spororeplicirajućih organizama što bi moglo objasniti zašto ti virusi nose jednolančanu DNK. Molekule DNK se kod različitih biljaka i životinja razlikuju po veličini. Najmanji broj nukleotida ima DNK virusa (samo nekoliko tisuća), molekula DNK bakterije sadrži nekoliko milijuna nukleotida, dok kod čovjeka taj broj prelazi nekoliko milijardi nukleotida.
Nukleinske kiseline prvi je opisao Friedrich Miescher 1869. godine, a prisutnost nukleinskih kiselina dokazano je 75 godina nakon toga, kad je otkrivena i njihova biološka funkcija. 30-ih i 40-ih godina smatralo se da su nositelji genetske informacije proteini. Prave naravi DNK opisao je 1928. godine Frederick Griffith, koji je otkrio transormirajući princip. To je otkrio pomoću pokusa s miševima s dvama tipova bakterija Diplococcus pneumoniae (R i S). U miševe je ubrizgao dva tipa bakterije D. pneumoniae, od kojih su neki bili živi, no oslabnjeni, tako da ne mogu prouzročiti bolest, a neke su bile posve mrtve (S). Od toga je većina miševa uginula. Otkriveno je da su mrtve bakterije na neki način transformirale žive, oslabljene bakterije. Godine 1953. Francis Crick i James Watson konstruirali su i opisali model prvog lanca DNK.
Modifikacije
Modifikacije baza

|

|

|
| Citozin | 5-metilcitozin | Timin |
  |

Ekspresija gena je zavisna od načina na koji je DNK pakovana u hromozomima, u strukturama zvanim hromatini. Modifikacije baza mogu da utiču na pakovanje. Regioni koji imaju nizak nivo ili odsustvo ekspresije obično sadrže visoke nivoe metilacije citozinskih baza.[204] Na primer, citozinska metilacija, proizvodi 5-metilcitozin, koji je važan za inaktivaciju X-hromozoma.[205]
Prosečni nivoi metilacije variraju između organizama. Crv Caenorhabditis elegans ne ispoljava citozinsku metilaciju,[206][207] dok kičmenjaci imaju visoke nivoe. Do 1% njihove DNK sadrži 5-metilcitozin.[208]
DNK metilacija je ključni deo normalnog razvoja organizma i ćelijske diferencijacije viših organizama. DNK metilacija stabilno menja obrasce genskog izražavanja u ćelijama tako da one mogu da „zapamte gde su bile“ ili da umanje ekspresiju gena. Na primer, ćelije programirane da budu Langerhansova ostrvca tokom embrionskog razvoja ostaju Langerhansova ostrvca tokom životnog veka organizma. DNK metilacija se tipično uklanja tokom formiranja zigota i ponovo uspostavlja tokom naknadnog ćelijskog razvoja. Nedavna istraživanja su pokazala da se metil grupe zapravo ne uklanjaju u zigotima, nego da dolazi do hidroksilacije metil grupa.[209] Neke metilacione modifikacije koje regulišu ekspresiju gena su nasledne i to se naziva epigenetičkom regulacijom. DNK metilacija supresuje izražavanje viralnih gena i drugih štetnih elemenata koji su bili inkorporirani u genom domaćina tokom vremena. DNK metilacija je isto tako osnova hromatinske strukture, koja omogućava ćelijama da poprime veliki broj karakteristika neophodnih za multicelularni život polazeći od jedne nepromenljive DNK sekvence.
DNK metilacija u poziciji 5 citozina ima specifičan efekat redukovanja genske ekspresije i nađena je kod svih kičmenjaka. U somatskom tkivu odraslih osoba, DNK metilacija se tipično javlja u CpG dinukleotidnom kontekstu, dok je u embrionskim matičnim ćelijama trend suprotan.[210][211][212]
DNK metilacija je od presudne važnosti u razvoju skoro svih tipova kancera.[213] Uprkos važnosti 5-metilcitozina, može doći do deaminacije čime se formira baza timin, tako da su metilisani citozini posebno skloni mutacijama.[214] Aberantni obrasci DNK metilacije su vezani za veliki broj ljudskih malignosti i grupišu se u dve distinktne forme: hipermetilacija i hipometilacija u odnosu na normalno tkivo. Hipermetilacija je jedna od glavnih epigenetičkih modifikacija koje represuju transkripciju putem promoterskog regiona tumor supresivnih gena.[215][216][217] Hipermetilacija se tipično javlja na CpG ostrvima u promoterskom regionu te proizvodi inaktivaciju gena. Globalna hipometilacija je bila implicirana u razvoj i progres kancera putem različitih mehanizama.[218][219][220]
Modifikacije drugih baza su metilacija adenina kod bakterija,[221][222] prisustvo 5-hidroksimetilcitozina u mozgu,[223] i glikozilacija uracila kojom se formira „J-baza“ u kinetoplastidima.[224][225]
Oštećenja

DNK može da bude oštećena mnogim vrstama mutagena, koji menjanju DNK sekvencu. Mutageni obuhvataju oksidacione agense,[229][230][231] alkilirajuće agense,[232][233] kao i elektromagnetnu radijaciju visoke energije, poput ultraljubičastog svetla[234][235] i X-zraka.[236][237] Tip proizvedenog DNK oštećenja zavisi od tipa mutagena. Na primer, UV svetlo može da ošteti DNK formiranjem timinskih dimera, koji su međusobno povezani između pirimidinskih baza.[238] S druge strane, oksidansi poput slobodnih radikala[239][240] ili vodonik peroksida[241][242] proizvode višestruke forme oštećenja, kao što su modifikacije baza, posebno guanozina, i prekidi dvostrukih lanaca.[243] Tipična ljudska ćelija sadrži oko 150.000 baza koje su podlegle oksidativnim oštećenjima.[244] Među tim oksidativnim ozledama, najopasniji su prekidi dvostrukih lanaca, jer se oni teško popravljaju i mogu da proizvedu genske mutacije, genetička umetanja i delecije iz DNK sekvence, kao i hromozomske translokacije.[245]
 |
Mnogi mutageni se uklapaju u prostor između dva susedna bazna para. To se naziva interkalacija.[246][247] Većina interkalatora su aromatični i planarni molekuli. Primeri su etidijum bromid,[248][249] akridini,[250][251] daunomicin,[252][253][254] i doksorubicin.[252][253][255] Da bi interkalator mogao da se uklopi između para baza, one se moraju razdvojiti. Stoga dolazi do distorzije DNK lanaca putem odvijanja dvostrukog heliksa. Time se inhibiraju transkripcija i replikacija DNK, što uzrokuje toksičnost i mutacije.[256] Rezultat je da DNK interkalatori mogu da budu karcinogeni, i u slučaju talidomida, teratogeni.[257][258] Drugi, poput benzo[a]piren diol epoksida[259][260] i aflatoksina,[261][262][263] formiraju DNK adukte koji indukuju greške u replikaciji.[264] Uprkos tome, usled njihove sposobnosti da inhibiraju DNK transkripciju i replikaciju, grupa sličnih toksina se takođe koriste u hemoterapiji za inhibiranje brzog rasta ćelija kancera.[265]
Mutacije

Mutacije mogu da uzrokuju dupliranje velikih delova DNK, obično putem genetičke rekombinacije.[266] Ta dupliranja su glavni izvor polaznog materijala za evoluciju novih gena. Od nekoliko desetina do nekoliko hiljada gena se duplira u životinjskom genomu svakih milion godina.[267] Većina gena pripada većim familijama gena sa zajedničkim nasleđem.[268] Novi geni nastaju na nekoliko načina. Do taga najčešće dolazi putem duplikacije i mutacije nasleđenih gena, ili rekombinacijom delova različitih gena čime se formiraju kombinacije sa novim funkcijama.[269][270]
Ovde, domeni deluju kao moduli, svaki od kojih ima specifičnu i nezavisnu funkciju. Njihove kombinacije mogu da proizvedu gene koji kodiraju nove proteine sa jedinstvenim osobinama.[271] Na primer, oči čoveka koriste četiri gena za formiranje struktura koje reaguju na svetlo: tri za raspoznavanje boja i jedan za noćni vid. Sva četiri su nastala od zajedničkog predačkog gena.[272] Još jedna prednost dupliranja gena (ili čak celokupnog genoma) je da to povećava redundantnost. Time se omogućava jednom genu da u paru poprimi novu funkciju, dok druga kopija ima originalnu funkciju.[273][274] Drugi tipovi mutacija ponekad kreiraju nove gene iz prethodno nekodirajuće DNK.[275][276]
Promene u broju hromozoma mogu da obuhvate mutacije još većih razmera, pri kojima se DNK segmenti hromozoma odvajaju i zatim preuređuju. Na primer, kod ranih hominina, dva hromozoma su spajanjem dala ljudski hromozom 2. Ta fuzija se nije javila u rodovima drugih čovekolikih majmuna, te su kod njih ta dva hromozoma zasebna.[277] U evoluciji, najvažnija posledica takvih hromozomska rearanžmana jeste ubrzanje divergencije populacija u nove vrste putem umanjivanja verovatnoće ukrštanja između populacija.[278]
Sekvence DNK koje mogu da se pomeraju u genomu, poput transpozona, sačinjavaju glavnu frakciju genetičkog materijala biljki i životinja, i smatra se da su bile važne u evoluciji genoma.[279] Na primer, više od milion kopija Alu sekvence je prisutno u humanom genomu, i te sekvence poseduju funkcije kao što je regulacija genske ekspresije.[280] Još jedan efekat tih mobilnih DNK sekvenci je da njihovo pomeranje unutar genoma može da prouzrokuje mutacije ili delecije postojećih gena, te one stoga doprinose genetičkoj raznovrsnosti.[281][282][283]
Neletalne mutacije se akumuliraju unutar genskog fonda i uvećavaju količinu genetičke varijabilnosti.[284] Obilje nekih genetičkih promena unutar genskog fonda može da bude redukovano prirodnom selekcijom, dok se druge „povoljnije“ mutacije mogu akumulirati i proizvesti adaptivne promene.
Na primer, leptir može da proizvede potomstvo sa novim mutacijama. Većina tih mutacija neće imati efekta, dok jedna može da promeni boju jednog od leptirovih potomaka, čineći ga teže (ili lakše) uočljivim za predatore. Ako je ta promena boje korisna, verovatnoća preživljavanja tog leptira i proizvođenja potomstva je u izvesnoj meri povećana, i tokom vremena broj leptira sa tom mutacijom može da formira veći udeo populacije.
Neutralne mutacije se definišu kao mutacije čiji efekti ne utiču na adaptivnu sposobnost jedne individue.[285][286] One mogu da se akumuliraju tokom vremena usled genetičkog drifta. Veruje se da ogromna većina mutacija nema značajan uticaj na adaptivnost organizma. Isto tako, mehanizmi DNK popravke su u stanju da poprave većinu promene pre nego što postanu stalne mutacije, i mnogi organizmi imaju mehanizme za eliminisanje inače permanentno mutiranih somatskih ćelija.
Korisne mutacije mogu da poboljšaju reproduktivni uspeh.[287][288]
Genetičke rekombinacije
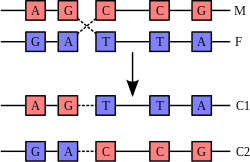

DNK heliks obično ne formira interakcije sa drugim DNK segmentima, i u ljudskim ćelijama različiti hromozomi čak zauzimaju zasebne oblasti jedra koje se nazivaju „hromozomske teritorije“.[291] Ova fizička separacija različitih hromozoma je važna za sposobnost DNK da funkcioniše kao stabilna riznica informacija. Jedan od retkih slučajeva kad hromozomi formiraju interakcije je tokom hromozomskog krosing-overa u procesu rekombinacije. Pri ukrštanju hromozoma dva DNK heliksa se raskidaju, zamenjuju sekcije i ponovo spajaju.
Rekombinacija omogućava hromozomima da razmene genetičke informacije i proizvedu nove kombinacije gena. Time se povećava efikasnost prirodne selekcije i ona može da bude važna za brzu evoluciju novih proteina.[292] Genetička rekombinacija može da bude deo popravke DNK, posebno u ćelijskom odgovoru na raskidanje dvostrukih lanaca.[293]
Najčešća forma hromozomskog krosing-overa je homologna rekombinacija, gde dva hromozoma razmenjuju veoma slične sekvence.[7][75] Nehomologna rekombinacija može da bude štetna za ćelije, jer ona može da proizvede hromozomske translokacije i genetičke abnormalnosti. Reakciju rekombinacije katalizuju enzimi poznati kao rekombinaze, poput RAD51.[294] Prvi stepen rekombinacije je raskidanje lanca dvostrukog heliksa bilo posredstvom endonukleaze ili usled oštećenja DNK.[295] Serija koraka koji su delom katalizovani rekombinazom dovodi do spajanja dva heliksa u najmanje jedan Holidejov spoj, u kome su segmenti jednog lanca svakog heliksa spojeni sa komplementarnim lancem drugog heliksa.[296][297] Holidejov spoj je tetraedralna struktura koja se može pomerati duž hromozoma, zamenjujući jedan lanac za drugi. Reakcija rekombinacije se zatim zaustavlja raskidanjem spoja i religacijom oslobođene DNK.[298]
U normalnoj mejozi svaka hromatida odlazi u poseban gamet. Gameti koji sadrže hromatide koje su razmenjivale delove nazivaju se krosing-over gameti. Jedinke koje nastaju od takvih gameta nazivaju se rekombinanti.[299][300]
Geni na jednom hromozomu nazivaju se vezani geni. Oni se zajedno prenose u potomstvo i da ne postoji krosing-over uvek bi se javljali u istim kombinacijama. Broj grupa vezanih gena jednog organizma jednak je njegovom broju hromozonih haploida. Zajedničko ispoljavanje dva ili više gena koji se nalaze na istom hromozomu naziva vezano nasleđivanje (korelativno nasleđivanje). U stvarnosti međutim, nije dovoljno da se dva gena nalaze na istom hromozomu da bi se vezano nasleđivali. Oni moraju biti vrlo blizu jedan do drugog na istom hromozomu. Ukoliko to nije slučaj može doći do njihovog rekombinovanja tokom krosing-overa. Verovatnoća odigravanja krosing-overa između dva gena na istom hromozomu zavisi od njihovog međusobnog rastojanja. Što je to rastojanje veće i verovatnoća da će doći do krosing-overa je veća i obratno. U genomu čoveka postoje geni između kojih je rastojanje toliko malo da se praktično krosing-over ne odigrava. Takvi skupovi gena koji se kao celina prenose na potomstvo nazivaju se haplotipovi.[7][75]
Činjenica da učestalost krosing-overa zavisi od rastojanja između gena koristi se prilikom mapiranja gena na hromozomu (određivanje mesta genima na hromozomu).[301][302] Genetičke mape koje se dobijaju na osnovu učestalosti krosing-overa daju nam uvid o relativnom položaju gena na hromozomima.[303][304][305] Rastojanje između dva gena procenjuje se na osnovu broja krosing-over gameta na 100 gameta (u %). Pri tome 1% krosing-overa predstavlja jedinicu rastojanja ili centimorgan (sM), tako da je 1 sM = 1% krosing-overa.[306][307][308]
Biološke funkcije
DNK se obično javlja u obliku linearnih hromozoma kod eukariota, i kružnih hromozoma kod prokariota. Set hromozoma u ćeliji čini njen genom. Ljudski genom ima oko 3 milijarde baznih parova DNK grupisanih u 46 hromozoma.[309] Informacije su sadržane u delovima sekvence DNK koji se nazivaju geni. Transmisija genetičke informacije sadržane u genima se ostvaruje putem komplementarnog sparivanja baza. Na primer, tokom transkripcije, kad ćelija koristi informaciju u genima, DNK sekvenca se kopira u komplementarnu RNK sekvencu putem privlačenja između DNK i korektnih RNK nukleotida. Obično se ova RNK kopija zatim koristi za pravljenje odgovarajuće proteinske sekvence u procesu translacije, koja zavisi od istih interakcija između RNK nukleotida. U alternativnom maniru, ćelija može da jednostavno kopira svoj genetički sadržaj u procesu replikacije DNK.
Geni i genomi
Genomska DNK je čvrsto i uredno upakovana procesom koji se zove DNK kondenzacija tako da se može smestiti u mali dostupni prostor unutar ćelije. Kod eukariota, DNK je locirana u ćelijskom nukleusu. Manje količine DNK su prisutne i u mitohondrijama i hloroplastima. Kod prokariota, DNK se nalazi unutar tela nepravilnog oblika u citoplazmi koje se naziva nukleoid.[310] Kompletna genetička informacija jednog organizma je njegov genotip. Gen je jedinica nasleđivanjai i region DNK koji proizvodi specifičnu karakteristiku organizma. Geni sadrže otvorene okvire čitanja[311][312] koji se mogu transkribovati, regulatorne sekvence kao što su promoteri, i pojačivače,[313] koji kontrolišu transkripciju otvorene okvire čitanja.
Kod mnogih vrsta, samo mala frakcija totalne sekvence genoma kodira proteine. Na primer, samo oko 1,5 % humanog genoma se sastoji od protein kodirajućih eksona, i preko 50% humane DNK su nekodirajuće ponavljajuće sekvence.[314] Razlozi za prisustvo tolike količine nekodirajuće DNK u eukariotskim genomima i izuzetno velike razlike u veličinama genoma, ili C-vrednostima, između vrsta predstavlja dugogodišnju zagonetku poznatu kao „enigma C-vrednosti“.[315] Međutim, DNK sekvence koje ne kodiraju proteine još uvek mogu da kodiraju funkcionalne nekodirajuće RNK molekule, koji učestvuju u regulaciji genske ekspresije.[316][317]
Neke nekodirajuće DNK sekvence imaju strukturne uloge u hromozomima. Telomere i centromere[318][319] tipično sadrže mali broj gena, ali su važni za funkciju i stabilnost hromozoma.[114][320] Bogata familija nekodirajuće DNK kod ljudi su pseudogeni,[268][321] koji su kopije gena koje su onesposobljene mutacijama.[322] Te sekvence su obično samo molekulski fosili, mada one mogu povremeno da služe kao sirovi genetički materijal za kreiranje novih gena putem procesa dupliranja gena[323][324] i divergencije.[325][326][327][328]
Transkripcija i translacija



Gen je DNK sekvenca koja sadrži genetičke informacije i koja može da utiče na fenotip organizma.[330][331] Struktura i/ili enzimska aktivnost svakog proteina prvenstveno proizlazi iz njegove primarne sekvence aminokiselina. Putem određivanja sekvence aminokiselina proteina geni imaju sposobnost nošenja informacije neophodne za definisanje aktivnog polipeptidnog lanca. Na taj način jedan jednostavan tip molekulske strukture ima sposobnost izražavanja bezbrojnih proteinskih formi. Kolektivno dejstvo raznih proteinskih proizvoda ćelije sprovodi katalitičke i strukturne aktivnosti koje su neophodne za uspostavljanje fenotipa.
Unutar gena, sekvenca baza duž DNK lanca definiše sekvencu informacione RNK, koja zatim definiše jednu ili više proteinskih sekvenci. Odnos između nukleotidnih sekvenci gena i aminokiselinskih sekvenci proteina je određen pravilima translacije, koja su poznata kao genetički kod. On se sastoji od reči sa tri slova koje se nazivaju kodoni. Oni su formirani od sekvenci sa tri nukleotida (npr. ACT, CAG, TTT).[332] Genetički kod dekodira kompleksni aparat koji stoji između nukleinske kiseline i proteina. Taj aparat je esencijalan za prenos informacije koju sadrži DNK. Samo jedan od dva DNK lanca kodira protein, tako da se genetički kod zapisuje kao sekvenca nukleotida, a ne baznih parova.
Tokom transkripcije, kodoni gena se kopiraju u informacione RNK molekule posredstvom RNK polimeraze. Te RNK kopije se zatim dekodiraju u ribozomima koji čitaju RNK sekvence putem baznog sparivanja informacione RNK sa transportnim RNK molekulima, koji nose aminokiseline. Pošto postoje četiri baze u kombinacijama od tri slova, moguća su 64 kodona (43 kombinacije).[333] Kodoni kodiraju dvadeset standardnih aminokiselina. Većina aminokiselina je kodirana sa više od jednog kodona. Postoje tri stop ili nesmisaona kodona koji označavaju kraj kodirajućeg regiona. To su: TAA, TGA i TAG kodoni.[334]
Gen sadrži seriju kodona koja se čita sekvencijalno od početne tačke na jednom kraju do krajnje tačke na drugom. Napisana u konvencionalnom 5'-3' smeru, sekvenca nukleotida DNK lanca koja kodira protein odgovara sekvenci aminokiselina napisanoj u pravcu od N-terminusa do C-terminusa.
Opšta baza koda je otkrivene putem genetičke analize mutacija rII regiona bakterijskog virusa, fag T4. Krik je 1961. pokazao da se kod mora čitati u nepreklapajućim tripletima počevši od fiksne početne tačke. Nepreklapanje znači da se svaki kodon sastoji od tri nukleotida i da su uzastopni kodoni predstavljeni uzastopnim trinukleotidima. Upotreba fiksne početna tačke znači da konstrukcija proteina mora da počne na jednom kraju i teče ka drugom, tako da se različiti delovi kodirajuće sekvence ne mogu nezavisno čitati.
Ako se genetički kod čita u nepreklapajućim tripletima, onda postoje tri moguća načina transliranja nukleotidne sekvence u protein u zavisnosti od početne tačke. Oni se nazivaju okvirima čitanja. Na primer za sekvencu ACGACGACGACGACGACG tri okvira čitanja su:
- ACG ACG ACG ACG ACG ACG
- CGA CGA CGA CGA CGA CGA
- GAC GAC GAC GAC GAC GAC
Mutacija koja umetne ili ukloni jednu bazu menja okvir čitanja celokupne sekvence. Promena te vrste se naziva pomeranje okvira (engl. frameshift). Pošto se sekvenca novog okvira čitanja kompletno razlikuje od stare, celokupna aminokiselinska sekvenca proteina je promenjena iza mesta mutacije, te se stoga funkcija proteina verovatno kompletno gubi.
Mutacije promene okvira čitanja mogu da indukuju akridini, jedinjenja koja se vezuju za DNK i izobličavaju strukturu dvostrukog heliksa, uzrokujući inkorporaciju ili izostavljanje dodatne baze tokom replikacije. Svaka mutacija uzrokovana akridinom dovodi do adicije ili uklanjanja jednog baznog para. Ako jedna akridinska mutacija proizvede na primer adiciju nukleotida, DNK se može vratiti u početno stanje uklanjanjem to nukleotida. Do reverzije se doći i odstranjivanjem različite baze na mestu u blizini prvog. Kombinacija takvih mutacija daje veoma korisne informacije o prirodi genetičkog koda.
Originalna analiza je izvedena genetičnim putem, tako što su sve akridinske mutacije klasifikovane u dve grupe, obeležene sa (+) i (-). Oba tipa mutacija uzrokuju frejmšift. Tip (+) putem adicije baze, a tip (-) putem delecije baze. Dvostruke kombinacije tipova (+ +) i (- -) su i dalje mutirane. Međutim, kombinacije tipova (+ -) i (- +) se međusobno poništavaju, te je jedan od mutanta supresor drugog. Ovi rezultati pokazuju da se genetički kod mora čitati u sekvenci sa okvirom čitanja koji ima fiksnu početnu tačku, tako da adicija i delecija mogu da kompenzuju jedna drugu, dok dvostruke adicije ili dvostruke delecije zadržavaju karakter mutanta. Ovi nalazi ne daju indikaciju o veličini kodona.
Kad se formiraju trostruke mutacije, samo (+ + +) i (- - -) kombinacije pokazuju početni fenotip, dok ostale kombinacije ostaju mutirane. Ako se pretpostavi da tri adicije ili tri delecije odgovaraju respektivno adiciji ili izostavljanju jedne aminokiseline, može se zaključiti da se kod čita u tripletima. Nekorektna aminokiselinska sekvenca se nalazi između dva spoljašnja mesta mutacija, dok sekvence na oba kraja ostaju nepromenjene.
Replikon
Bilo da ćelija ima samo jedan hromozom (kao kao prokariota) ili mnoštvo hromozoma (kao kod eukariota), celokupni genom se mora replikovati jednom tokom svake ćelijske deobe.[3] Dva opšta principa se koriste za poređenje stanja replikacije sa uslovima ćelijskog ciklusa:
- Inicijacija DNK replikacije obavezuje ćeliju da izvrši podelu. Sa te tačke gledišta broj potomaka ćelije je određen serijom odluka o inicijaciji replikacije DNK.
- Ako je replikacija u toku, podela nije dozvoljena dok se replikacioni čin ne završi. Završetak replikacije može da proizvede signal za sledeću ćelijsku podelu. Duplirani genomi se razdvajaju u dve ćelije ćerke (putem mitoze kod eukariota). Jedinica segregacije je hromozom.
Regulatorni geni ćelijskog ciklusa aktiviraju prekidače koji iniciraju DNK replikaciju i pokreću samu deobu. Kod prokariota, inicijacija replikacije je događaj u kome učestvuje jedinstveno mesto bakterijskog hromozoma, i proces deobe je praćen razvojem pregrade. Kod eukariotskih ćelija, inicijacija replikacije je identifikovana početkom S faze, dužeg perioda tokom kojeg dolazi do sinteze DNK, i koji obuhvata mnoge individualne inicijacione događaje. Čin podele se ostvaruje reorganizacijom ćelije tokom mitoze.
Jedinica DNK u kojoj se javlja individualni čin replikacije se naziva replikon. Svaki replikon se aktivira jednom i samo jednom u svakom ćelijskom ciklusu. Replikon je definisan posedovanjem kontrolnih elemenata potrebnih za replikaciju. On sadrži mesto početka na kome se inicira replikacija. On isto tako može da sadrži „terminus“ na kome se replikacija zaustavlja. Svaka sekvenca vezana za mesto početka, ili preciznije sekvenca koja nije razdvojena od mesta početka terminusom, se replikuje kao deo replikona. Mesto početka može da utiče samo na DNK molekul na kome se nalazi. Genom prokariotske ćelije je jedan replikon, tako da su jedinice replikacije i segregacije na istom mestu. Najveći takav replikon je sam bakterijski hromozom. Replikon je fleksibilna jedinica. U slučaju bakterijskih hromozoma, on se koristi za formiranje kopija dupliranjem dvolančane DNK. On se takođe može koristiti za generisanje jednolančanih kopija genoma faga ili plazmida u monometrijskim ili multimetrijskim formama. Mod reprodukcije replikona zavisi od prirode interakcija koje se javljaju tokom inicijacije na mestu početka. Opšti princip je da je replikacija kontrolisana stupnjem inicijacije. Nakon početka replikacije, ona se nastavlja dok se celokupni genom ne duplira.[1]
Glavna razlika u organizaciji bakterijskih i eukariotskih genoma je u njihovoj replikaciji. Svaki eukariotski hromozom sadrži veliki broj replikona, tako da jedinica segregacije obuhvata mnoštvo jedinica replikacije. Time se dodaje nova dimenzija problemu kontrole. Svi replikoni na hromozomu se moraju aktivirati tokom ćelijskog ciklusa, mada oni nisu istovremeno aktivni, nego se aktiviraju tokom dužeg perioda. Svaki replikon se mora aktivirati samo jednom u datom ćelijskom ciklusu. Signal mora da razlikuje replicirane od nerepliciranih replikona, tako da do aktivacije replikona dođe samo jednom. Pošto su mnogi replikoni nezavisno aktivirani, mora da postoji signal koji označava da je celokupan proces replikacije završen.
Replikacija može da bude jednosmerna i dvosmerna. Tip je određen time da li se jedna ili dve replikacione račve formiraju. Kod jednosmerne replikacije, jedna replikaciona viljuška napušta mesto početka i kreće se duž DNK. Kod dvosmerne replikacije, dve replikacione viljuške se formiraju i kreću se u suprotnim smerovima. Kad se replikacija DNK posmatra pod elektronskim mikroskopom, replikacioni region izgleda kao oko unutar nereplikovane DNK. Međutim na osnovu izgleda segmenta se ne može reći da li je replikacija jednosmerna ili dvosmerna. Oko može da predstavlja bilo koju od te dve strukture. Ako je oko formirano jednosmernom replikacijom, ono predstavlja nepomerno mesto početka i pokretnu replikacionu račvu. Ako je formirano dvosmernom replikacijom, ono predstavlja par replikacionih račvi. Nezavisno od tipa, progres replikacije proširuje oko dok se ultimatno ne proširi na ceo replikon. Kad je replikon kružan, prisustvo oka formira θ strukturu.
Broj replikacionih račvi replikacionog oka se može odrediti na dva načina. Izbor metoda zavisi od toga da li se analizira definisani molekul DNK ili nedefinisani region ćelijskog genoma. U slučaju definisanog molekula, može se koristiti elektronska mikroskopija za merenje rastojanja krajeva replikacionog oka od krajeva DNK. Zatim se pozicija svakog kraja oka može porediti u molekulima sa koji imaju različite veličine oka. Ako je replikacija jednosmerna samo se jedan kraj pomera. Ako je replikacija dvosmerna oba kraja se pomeraju, dok je mesto početka u sredini. U slučaju nedefinisanih regiona velikog genoma, dva uzastopna radioaktivna pulsa se mogu koristiti za obeležavanje kretanja replikacione viljuške. Ako jedan od pulseva ima intenzivniju oznaku, oni se mogu razlikovati po relativnim intenzitetima oznaka primenom autoradiologije. Kod jednosmerne replikacije jednoj oznaci sledi druga na jednom kraju oka, dok se kod dvosmerne javlja simetričan obrazac na obe strane oka. Simetrični obrazac se obično uočava u replikonima eukariotskih hromozoma.
Savremeniji metod mapiranja mesta početka sa povećanom rezolucijom koristi promenljive efekte promene oblika putanja nakon elektroforetske migracije DNK. Tehnika dvodimenzionog mapiranja razdvaja restrikcione fragmente replikujuće DNK elektroforezom po masi u prvoj dimenziji, dok je u drugoj dimenziji kretanje prvenstveno određeno oblikom. Različiti tipovi replikacionih molekula slede karakteristične puteve, koji se mere njihovom devijacijom od linije koju bi sledili linearni molekuli udvostručene veličine. Jednostavna Y-struktura u kojoj se jedna viljuška kreće duž linearnog fragmenta sledi kontinuirani put. Tačka inflekcije se javlja kad su sve tri grane jednake dužine. Ta struktura maksimalno odstupa od linearne DNK. Analognim razlozi određuju puteve dvostrukih Y-struktura ili mehura. Asimetrični mehur sledi isprekidani put, koji se prekida u tački u kojoj se mehur konvertuje u Y-strukturu.
Bakterijski replikon
Bakterijski replikon treba da podržava sledeće funkcije da bi se korektno nasleđivao
- Inicijacija replikacionog ciklusa
- Kontrola frekvencije inicijalnih događaja
- Segregacija replikovanih hromozoma u ćelije ćerke
Prve dve funkcije su osobine mesta početka. Segregacija može da bude nezavisna funkcija, ali u prokariotskim sistemima obično počiva na sekvenci u blizini mesta početka. To nije slučaj kod eukariota. DNK sekvenca mesta početka replikona ima sposobnost podržavanja replikacije bilo koje DNK sekvence u koju se unese. Kad se ona klonira u molekul koji ne sadrži mesto početka, rekonstrukcija formira plazmid koji ima sposobnost autonomne replikacije. Mesta početka su identifikovana kod bakterija, kvasaca, hloroplasta i mitohondrija. Opšte svojstvo je da je sekvenca bogata AT parovima. Smatra se da je to usled potrebe da se dvolančana DNK otopi kako bi se inicirala replikacija.
Genom Ešerihije koli se dvosmerno replikuje iz jednog mesta početka, koje se identifikuje kao genetički lokus oriC.[335][336] Dodatak oriC segmenta u bilo koju DNK sekvencu formira veštački plazmid koji se može replikovati u E. Coli. Putem redukovanja veličine kloniranog fragmenta oriC utvrđeno je da se region neophodan za iniciranje replikacije sastoji od 245 bazna para. Plazmidi koji se korektno iniciraju mogu da imaju iregularnu segmentaciju, ali se to može stabilizovati uvođenjem dodatnih sekvenci. Iz toga sledi da mesto početka koje je neophodno za inicijaciju ne sadrži dovoljno informacija da omogući podelu dupliranih DNK molekula u ćelije ćerke pri podeli bakterije. Funkcije koje učestvuju u podeli se mogu identifikovati karakterizacijom sekvenci koje uslovljavaju segregacionu stabilnost plazmida.
Prokariotski replikoni su obično kružni. Kružne strukture obuhvataju bakterijski hromozom, plazmide i mnoge bakteriofage. One su isto tako česte u hloroplastima i mitohondrijskim DNK molekulima. Replikacija kružnih molekula izbegava problem replikacije krajeva, ali ima problem završavanja replikacije. Bakterijski hromozom se dvosmerno replikuje kao jedna jedinica počevši od oriC lokacije. Dve replikacione račve se kreću oko genoma aproksimativno istom brzinom do tačke sastajanja, i do terminacije dolazi u diskretnom regionu. Nakon terminacije same replikacije DNK, enzimi višeg nivoa koji manipulišu strukture su neophodni da bi se dva novonastala hromozoma razdvojila. Sekvence koje uzrokuju terminaciju se nazivaju ter mesta. Ona se sastoje od kratke (~23 bazna para dugačke) sekvence koja uzrokuje terminaciju in vitro. Terminacione sekvence funkcionišu samo u jednom smeru. Terminacija zahteva protein kodiran TUS genom[337][338] koji prepoznaje konsenzus sekvencu i sprečava dalji napredak replikacione viljuške.
Kod Ešerihije koli replikaciona račva se obično zaustavlja u tački na pola puta oko hromozoma. Postoje dva terminaciona regiona (terD,A i terC,B) locirana oko 100 kb na svakoj strani mesta sretanja. Svaki terminalni region je specifičan za jedan smer kretanja račve, i oni su raspoređeni na takav način da bi svaka račva morala da pređe drugi završni region da bi dosegla do završnog kraja koji prepoznaje. Ovaj aranžman formira klopku za replikacione račve. Ako je iz bilo kog razloga jedna račva kasni, tako da račve ne uspeju da se sretnu u uobičajenoj centralnoj poziciji, brža račva će biti zaustavljena u ter regionu i čekaće dolazak sporije račve.
U slučaju da replikaciona račva (koja se kreće deset puta brže) naiđe na RNK polimerazu koja se kreće u istom smeru, ona je obilazi bez poremećaja transkripcije. Mehanizam ove interakcije nije poznat. U slučaju da se RNK polimeraza kreće u suprotnom smeru, konflikt se verovatno ne bi mogao rešiti, te može doći do letalnog ishoda. To je mogući razlog što su kod Ešerihije koli skoro sve aktivne transkripcione jedinice orijentisane tako da se izražavaju u istom smeru kao i replikaciona račva. Izuzeci su jedino male transkripcione jedinice koje se retko izražavaju.
Eukariotski replikon
Kod eukariotskih ćelija, replikacija DNK je ograničena na deo ćelijskog ciklusa. S faza se javlja kao deo interfaze koja obično traje nekoliko časova kod ćelija viših eukariota. Replikacija velike količine DNK sadržane u eukariotskom hromozomu se ostvaruje podelom u mnoštvo replikona. Samo deo tih replikona učestvuje u replikaciji u bilo kojem trenutku S faze. Svaki replikon se aktivira u specifično vreme, mada evidencija o tome nije potpuna. Signal za početak S faze je aktivacija prvog replikona. Tokom sledećih nekoliko sati dolazi do aktivacije drugih replikona. Kontrola S faze stoga obuhvata dva procesa: izlazak ćelije iz prethodne G1 faze, i inicijaciju replikacije individualnih replikona na uređen način.
Najveći deo poznatih svojstava pojedinačnih replikona je dobijen putem autoradiografskih studija. Hromozomski replikoni obično imaju dvosmernu replikaciju. Poteškoća u karakterisanju pojedinačnih jedinica je u tome da se susedni replikoni spajaju i proizvode velike replikacione mehure. Pristup koji se koristi za razlikovanje individualnih replikona od spojenih se obično oslanja na segmente DNK u kojima se može videti nekoliko aktivnih replikona, koji su verovatno aktivirani u približno isto vreme i čije račve se još nisu srele. Postoje dokazi da „regionalna“ kontrola može da proizvede izvestan oblik regulacionog obrasca u kome su grupe replikona inicirane više ili manje koordinirano, što je u suprotnosti sa mehanizmom u kome su individualni replikoni aktivirani jedan po jedan u rasutim oblastima genoma. Dve strukturne osobine sugerišu mogućnost organizacije na velikoj skali. Veoma veliki regioni hromozoma se mogu karakterisati kao „rano replikujući“ ili „kasno replikujući“, iz čega sledi da postoji izvesna raspodela između replikona koji se aktiviraju rano i kasno. Vizuelizacija replikacionih račvi obeleženih sa DNK prekursorima pokazuje 100-300 centara, umesto uniformne raspodele. Svaki centar verovatno sadrži >300 replikacionih račvi. Račve mogu da predstavljaju fiksne strukture kroz koje replikujuća DNK mora da prođe.
| Eukariotski replikoni su mali i replikuju se sporije od bakterijske DNK | |||
|---|---|---|---|
| Organizam | Broj replikon (bp) | Prosečna dužina | Brzina kretanja (bp/min) |
| Bakterija | 1 | 4.200 | 50.000 |
| Kvasac | 500 | 40 | 3.600 |
| Voćna mušica | 3.500 | 40 | 2.600 |
| Žaba | 15.000 | 200 | 500 |
| Miš | 25.000 | 150 | 2.200 |
| Biljka | 35.000 | 300 | |
U grupama aktivnih replikona, prosečna veličina jedinice se meri rastojanjem između mesta početaka. Brzina kretanja replikacione račve se može proceniti iz maksimalnog rastojanja koje autoradiografski trag pređe tokom datog vremenskog intervala. Pojedinačni eukariotski replikoni su relativno mali, iako njihova dužina varira više od deset puta unutar genoma. Brzina kojom se oni replikuju je znatno manja od brzine bakterijske replikacije. Genom sisara bi se mogao replikovati u toku jednog sata, ako bi svi replikoni simultano funkcionisali. Međutim, S faza zapravo traje duže od šest sati u tipičnim somatskim ćelijama, iz čega sledi da je do 15% replikona aktivno u bilo kom momentu. Postoje neki izuzetni slučajevi, kao što je rani embrionski razvoj Drozofila embriona, gde je dužina S faze kompresovana simultanim funkcionisanjem velikog broja replikona.
Dostupna evidencija sugeriše da hromozomski replikoni nemaju terminuse na kojima se replikacione račve zaustavljaju i enzimski kompleks disocira od DNK. Verovatniji scenario je da replikacione račve nastavljaju kretanje od svog mesta početka dok se ne sretnu sa račvom koja se kreće u suprotnom smeru.
Svaki DNK segment koji sadrži mesto početka bi trebalo da ima sposobnost replikacije. Mada su plazmidi retki kod eukariota, moguće ih je formirati putem podesnih manipulacija in vitro. To je ostvareno kod kvasca, mada ne i kod viših eukariota. Saccharomyces cerevisiae mutanti se mogu transformisati do divljeg tipa dodatkom DNK koja sadrži kopiju gena divljeg tipa. Neki DNK fragmenti kvasca (često kružni) imaju sposobnost veoma efektivnog transformisanja defektivnih ćelija. Ti fragmenti mogu da opstanu u ćelijama u neintegrisanom (autonomnom) stanju, poput samoreplikujućih plazmida. Fragmenti koji se transformišu sa visokom frekvencijom poseduje sekvence koje imaju sposobnost efektivne replikacije u kvascu. Taj segment se naziva ARS (autonomno replikujuća sekvenca). ARS elementi su izvedeni iz autentičnih mesta početka replikacije hromozoma. Sekvence sa ARS funkcijom se javljaju sa skoro jednakom frekvencijom kao i mesta početka replikacije. ARS elementi su sistematski mapirani na dužim regionima hromazoma. Samo deo njih se zapravo koristi za inicijaciju replikacije. Drugi su nemi, ili se možda koriste povremeno. Ako je tačno da neka mesta početka imaju varirajuću verovatnoću aktivacije, sledi da granice replikona nisu fiksne. U tom slučaju dati region hromozoma može da bude replikovan iz različitih mesta početaka u različitim ćelijskim ciklusima. ARS element se sastoji od AT bogatog regiona koji sadrži određena diskretna mesta na kojima mutacije imaju znatan uticaj. Sadržaj baza umesto same sekvence može da bude značajan za ostatak regiona.
Replikacija DNK molekula


Replikacija DNK molekula je veoma složen i važan proces. Stoga je puno vremena i truda je uloženo u njegovo razumevanje.
Ćelijska deoba je esencijalna za rast organizma. Tokom deobe ćelija dolazi do replikacije DNK tako da svaka od novonastalih ćelija ima isti genetički sadržaj kao i njihov roditelj. Dvolančana struktura DNK omogućava jednostavan mehanizam za replikaciju. Ovde se dva lanca razdvajaju i formira se komplementarna sekvenca za svaki od njih posredstvom enzima DNK polimeraza.[13] Taj enzim formira komplementarni lanac tako što nalazi korektnu bazu putem sparivanja komplementarnih baza, i njihovog vezivanja na originalni lanac. Pošto DNK polimeraze mogu da produže jedino DNK lanac u 5′ ka 3′ smeru, drugi mehanizmi se koriste za kopiranje antiparalelnih lanaca dvostrukog heliksa.[339] Na taj način, baze starog lanca određuju u kojoj sekvenci se baze pojavljuju u novom lancu, i ćelija dobija perfektnu kopiju svoje DNK.
Replikacija DNK se takođe može izvoditi in vitro (veštački, izvan ćelije). DNK Polimeraze, izolovane iz ćelija, i veštački DNK prajmeri se koriste za iniciranje sinteze DNK na poznatim sekvencama molekulskih templeta. Polimerazna lančana reakcija (PCR) je uobičajena laboratorijska tehnika u kojoj se primenjuje takva veštačka sinteza u cikličnom režimu radi umnožavanja specifičnog ciljnog DNK fragmenta iz DNK smeše.
Eukariotska replikacija
| DNK polimeraza | |||||||||
|---|---|---|---|---|---|---|---|---|---|

| |||||||||
| 3D struktura heliks-zavoj-heliks motiva DNK vezivanja ljudske DNK polimeraze beta (7ICG)[340] | |||||||||
| Identifikatori | |||||||||
| EC broj | 2. 7. 7.7 | ||||||||
| CAS broj | 9012-90-2 | ||||||||
| IntEnz | 7. 7.7 IntEnz view | ||||||||
| BRENDA | 7. 7.7 BRENDA entry | ||||||||
| ExPASy | 7. 7.7 NiceZyme view | ||||||||
| KEGG | 7. 7.7 KEGG entry | ||||||||
| MetaCyc | 7. 7.7 metabolic pathway | ||||||||
| PRIAM | 7. 7.7 profile | ||||||||
| PDB | 7. 7.7 RCSB PDB 7. 7.7 PDBe 7. 7.7%27 PDBj 7. 7.7 PDBsum | ||||||||
| Ontologija gena | AmiGO / EGO | ||||||||
| |||||||||
Replikacija DNK molekula počinje na mestu koji se zove oriC lokus.[335][336] Protein DNK-A se vezuje za oriC lokus i pritom se vrši hidroliza adenozin trifosfata. Ovo prvo nadovezivanje dovodi do početnog odvijanja DNK molekula iz spirale u dva linearna lanca povezana vodoničnim vezama. Da bi replikacija bila uspešna DNK mora da postane linearna, a ne spiralno uvijena, dakle mora da izgleda kao merdevine. Enzimi koji odvijaju DNK molekul u oblik merdevine se zovu helikaze.[341][342] Enzimi odvijaju DNK molekul veoma brzo, čak 75 do 100 revolucija u sekundi.[343] Ovakvo brzo odvijanje molekula DNK može da dovede do stvaranja tenzija polinukleotidnih lanaca. Ova pojava tenzija se na primer može videti kada se uviju pertle i kada pokušamo brzo da ih razdvojimo, pertle se uviju u čvorove usled tenzije. Da bi se ovo izbeglo stvaranje čvorova koje bi moglo da oštetiti DNK molekul, prisutni su enzimi koji se zovu DNK topoizomeraze. Oni popuštaju vodonične veze kako bi se tenzija i stvaranje čvorića izbeglo.[57][58][59] U isto vreme dok se DNK molekul razdvaja u oblik merdevina, struktura koja se naziva replikaciona viljuška (ili račva)[342] ide odmah iza topoizomeraza i razdvaja vodonične veze između parova (A-T i G-C). Da bi ovi polinukleotidni lanci ostali razdvojeni razdvajajući proteini se vezuju na obe strane svakog lanca i na taj način održavaju lance odvojene. Replikacija DNK molekula se može uporediti sa rajsferšlusom. Kada želimo da otvorimo rajsferšlus, vučemo mehanizam na dole, i na taj način dobijamo dve strane rajsferšlusa za razdvojenim zupčanicima. Na isti način se DNK razdvaja, pri čemu mehanizam rajsferšlusa predstavlja replikacionu viljušku.
Nakon razdvajanja postoje dva polinukleotidna lanca, jedan ide u pravcu 3'→ 5' dok drugi ide u pravcu 5'→ 3' (antiparalelnost). Veoma važan enzim koji sintetiše nove polinukleotidne lance DNK polimeraza δ,[344][345] može da sintetiše novi lanac samo u pravcu 5'→ 3'. To nije problem za vodeći lanac koji se sintetiše u pravcu kretanja replikacione viljuške.[346][347]
Sintezu oba lanca obavlja DNK polimeraza tek pošto se veže za roditeljski lanac koji služi kao matrica. Ovaj enzim ne može da se veže za ogoljeni lanac-matricu već zahteva postojanje začetnika (prajmera). Začetnik je kratki lanac RNK i njegovu sintezu katalizuje enzim primaza. Kada se kratki lanac RNK komplementarno spari (hibridizuje) sa početkom lanca matrice to omogućuje vezivanje DNK polimeraze i počinje sinteza novog lanca. Za sintezu lanca koji zaostaje potrebno je da se sintetiše veći broj začetnika. Okazakijeve fragmente, po završetku sinteze, međusobno povezuje enzim ligaza.[348][349]
Lanac koji se sintetiše pravcu suprotnom od pravca kretanja replikacione viljuške 3' → 5' ne može da bude sintetisan bez prekida. On se sintetiše u fragmentima koji se nazivaju Okazakijevi Fragmenti[350][351] (nazvanim po naučniku Reiji Okazaki koji je prvi ukazao na njihovo postojanje 1966.[352]) i pojavljuju se samo na lancu koji ide u ovom pravcu. Oni su komplementarni sa templetom zaostajućeg lanca, sa kojim formiraju dvolančane DNK sekcije. Okazakijevi fragmenti su između 100 do 200 nukleotida dugački kod eukariota, dok kod Ešerihije koli imaju 1 000 to 2 000 nukleotida. Oni su razdvojeni RNK primerima od ~10-nukleotida i do spajanja dolazi nakon uklanjanja prajmera.
Da bi novi DNK molekul bio kompletan i bez prekida, enzim ligaza ima ulogu lepka i vezuje fragmente jedan za drugi, i tako nastaju od jednog DNK molekula, dva novoformirana DNK molekula. DNK polimeraza β ima važnu ulogu u proveri novih DNK molekula,[353][354] tako što ide duž celih novonastalih lanaca, čita ih i proverava da li su sve baze korektno povezane (A-T i G-C). DNK replikacija se zaustavlja kada replikaciona viljuška naiđe na sekvencu na DNK molekulu koji kodira za stopiranje DNK replikacije.
Proces DNK replikacije je veoma komplikovan. Jedan od razloga za ovu kompleksnost je da novonastali DNK molekuli moraju da budu tačni. Greške u sintezi DNK molekula mogu da dovode do raznih bolesti i često su fatalne. Ovaj proces zvuči veoma neverovatno kada se uzme u obzir da se novih 850 baznih parova kod prokariota sintetiše u roku od jedne sekunde, dok je kod eukariota ova brzina je nešto niža, oko 150 baznih parova u jednoj sekundi.
Prokariotska replikacija
| Bac_DnaA_C | |
|---|---|
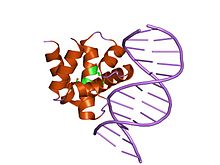
| |
| Kristalna struktura DnaA domena u kompleksu sa DNK kutijom (1J1V)[355] | |
| Identifikatori | |
| Simbol | Bac_DnaA_C |
| Pfam | PF08299 |
| Pfam klan | CL0123 |
| InterPro | IPR013159 |
| SKOP | 1j1v |
Prokariotska replikacija DNK je bidirekciona i započinje u centru replikacije (OriC).[356]
Inicijaciju DNK replikacije posreduje DnaA[357], protein koji se vezuje za region centra replikacije poznat kao DnaA kutija. Kod E. coli postoji pet DnaA kutija, svaka od kojih sadrži devet visoko konzerviranih baznih parova, konsenzus sekvencu 5' - TTATCCACA - 3'.[358] Posledica vezivanja DnaA za taj region je da DNK postaje negativno supernamotana. Nakon toga, regioni OriC ispred DnaA kutija (poznati kao DnaB kutije) se otapaju. Postoje tri takva regiona, i svaki je dugačak 13 baznih parova, i bogat AT parovima (što olakšava topljenje jer je manje energije potrebno za razlaganje dve vodonične veze. Taj region ima konsenzus sekvencu 5' - GATCTNTTNTTTT - 3.[359] Za rastapanje DnaB kutija je neophodan adenozin trifosfat (koji hidrolizuje DnaA). Nakon rastapanja, DnaA regrutuje heksamernu helikazu (šest DnaB proteina) na suprotne krajeve rastopljene DNK. Na tom mestu se formira replikaciona viljuška. Za regrutovanje helikaze je neophodno šest DnaC proteina, svaki od kojih se vezuje za jednu podjedinicu helikaze. Nakon formiranja tog kompleksa, dodatnih pet DnaA proteina se vezuje. DnaC se zatim odvaja, i kompleks je kompletan. Da bi se DNK replikacija nastavila potreban je jednolančani protein koji sprečava jednolančane DNK lance da formiraju sekundarnu strukturu, kao i za sprečavanje njihovog ponovnog međusobnog sparivanja. DNK giraza[360][361] je potrebna da bi se umanjio stres uzrokovan formiranjem negativnih supernamotaja formiranih posredstvom DnaB helikaze.[362][363] Odmotavanje DNK DnaB helikazom omogućava primazi (DnaG[364][365]) polimerazi da formira prajmer na svakom DNK templetu tako da DNK sinteza može da počne.
Nakon formiranja prajmera, DNK polimeraza III holoenzim započinje replikaciju DNK.[366][367] Njen katalitički mehanizam obuhvata upotrebu dva metalna jona u aktivnom mestu. Ovaj enzim ima sposobnost razlikovanja dezoksiribonukleotida i ribonukleotida. Metalni joni su generalno divalentni katjoni koji pomažu 3' OH da inicira nukleofilni napad na alfa fosfat dezoksiribonukleotida i da orijentiše i stabilizuje negativno naelektrisani trifosfat na dezoksiribonukleotidu. Nukleofilni napad 3' OH-a na alfa fosfat oslobađa pirofosfat, koji se naknadno hidrolizuje (neorganskom fosfatazom) u dva fosfata. Ova hidroliza završava sintezu DNK.
Terminacija replikacije DNK kod Ešerihije koli se obavlja putem upotrebe terminacionih sekvenci i Tus proteina.[337][338] Te sekvence omogućavaju da dve replikacione viljuške idu samo u jednom pravcu. Replikacija DNK inicijalno proizvodi dva povezana kružna DNK dupleksa, svaki od kojih se sastoji od jednog roditeljskog lanca i novoformiranog lanca. Kod Ešerihije koli topoizomeraza IV razdvaja kružne DNK duplekse.[368][369]
Fagne strategije
Neki bakteriofagovi imaju samo jednu strategiju opstanka. Nakon infekcije podložnog domaćina, oni menjaju njegove ćelijske funkcije i podređuju ih proizvodnji velikog broja virusnih potomaka. Rezultat litičke infekcije je smrt bakterijskog domaćina. U tipičnom litičkom ciklusu DNK (ili RNK) faga ulazi u ćeliju domaćin, njegovi geni se transkribuju u određenom redu, genetički materijal faga se replikuje, i proteinske komponente virusa se proizvode. Konačno, bakterija domaćin se otvara (lizira) da bi se oslobodilo formirano potomstvo faga.
Drugi fagovi imaju dvostruki način postojanja. Oni mogu da proizvode litičke cikluse, što je otvorena strategija kojom se proizvodi što više kopija za što kraće vreme. Oni isto tako imaju alternativnu formu postojanja, u kojoj je genom faga prisutan u bakteriji u latentnoj formi poznatoj kao profag. Ta forma propagacije se naziva lizogenija. U lizogenoj bakteriji profag je integrisan u bakterijski genom, i nasleđuje se na isti način kao i bakterijski geni. Usled posedovanja profaga, lizogena bakterija je imuna na dalju infekciju drugim fagovima istog tipa. Imunost se uspostavlja integracijom jedne kopije profaga, tako da bakterijski genom sadrži samo jednu kopiju profaga bilo kog tipa. Tranzicija se javlja između lizogenog i litičkog moda postojanja. Kad je fag proizveden litičkim ciklusom uđe u novu bakterijsku ćeliju, on bilo ponavlja litički ciklus ili ulazi u lizogeno stanje. Ishod zavisi od uslova infekcije i genotipa faga i bakterije. Profag se oslobađa lizogenih ograničenja procesom indukcije, u kome se izdvaja iz bakterijskog genoma, da formira slobodnu DNK faga, koja zatim prolazi kroz litički put. Alternativne forme u kojima se fagovi propagiraju su određene regulacijom traskripcije. Lisogenija se održava putem interakcije fagnog represora sa operatorom. Za litički ciklus je neophodna kaskada transkripcione kontrole. Tranzicija između dva načina života se ostvaruje uspostavljanjem represije (litički ciklus u lizogeniju), ili oslobađanjem od represije (indukcija lizogena u litički ciklus).
Plazmidi su još jedan tip postojanja DNK unutar bakterije. Oni su autonomne jedinice koje postoje u ćeliji kao ekstrahromozomni genomi. Plazmidi su samoreplikujući kružni DNK molekuli, koji se održavaju u ćeliji sa stabilnim i karakterističnim brojem kopija.[39] Drugim rečima, njihov broj ostaje konstantan iz generacije u generaciju. Neki plazmidi takođe imaju alternativne životne stilove. Oni mogu da postoje bilo u autonomnom ekstrahromozomskom stanju, ili mogu da budu umetnuti u bakterijski hromozom, i onda su nošeni kao njegov deo poput bilo kojeg dela sekvence. Takve jedinice se nazivaju epizomi, mada se termini plazmid i epizom često koriste kao sinonimi. Poput lizogenih fagova, plazmidi i epizomi održavaju sebičnu posedovanje njihove bakterije i često onemogućavaju drugim elementima istog tipa da se uspostave. Taj efekat se naziva imunost, mada se baza plazmidne imunosti razlikuje od lizogene imunosti. Neki plazmidi i epizomi se prenose između ćelija putem konjugativnog procesa koji obuhvata direktni kontakt između ćelija donora i primaoca.
Litički razvoj
Genomi faga su neophodno mali.[40] Kao i kod svih virusa, oni su ograničeni potrebom da se upakuje nukleinska kiselina unutar proteinskog omotača. To ograničenje diktira mnoge viralne reprodukcione strategije. Tipično virus preuzima kontrolu nad ćelijom domaćina i koristi njenu strukturu za replikaciju i izražavanje svojih gena. Obično fag sadrži gene čija funkcija je osiguravanje preferentne replikacije fagne DNK. Proteini kodirani tim genima obavljaju inicijaciju replikacije. Fag može da sadrži gen DNK polimeraze. Fag menja kapacitet ćelije domaćina da obavlja transkripciju. To se postiže zamenom RNK polimeraza ili modifikovanjem sposobnosti inicijacije ili terminacije. Rezultat je uvek isti: fagni iRNK molekuli su preferentno transkribovani. U pogledu sinteze proteina fag se obično oslanja na aparat domaćina, preusmeravajući njegove aktivnosti zamenom bakterijske iRNK fagnom.
Litički razvoj se ostvaruje biohemijskim putem u kome se fagni geni izražavaju u specifičnom redosledu. Time se osigurava da je odgovarajuća količina svake komponente prisutna u odgovarajuće vreme. Ciklus se može podeliti u dva opšta puta:
- Rana infekcija, koja je period od ulaza DNK u ćeliju do početka replikacije
- Kasna infekcije je period od početka replikacije do krajnjeg koraka liziranja ćelijskog zida da bi se oslobodilo fagno potomstvo.
Rana faza je posvećena proizvodnji enzima koji učestvuju u reprodukciji DNK. To obuhvata enzime DNK sinteze, rekombinacije, i ponekad modifikacije. Njihove aktivnosti uzrokuju akumulaciju rezervoara genoma faga. U tom rezervoaru genomi se konstantno replikuju i rekombinuju. Tokom kasnije faze proteinske komponente se sintetišu. Često je neophodno mnoštvo različitih proteina da bi se formirale strukture glave i repa, tako da se najveći deo genoma faga sastoji od strukturnih gena. Osim strukturnih proteina potrebni su „montažni proteini“ koji pomažu u konstruisanju čestica fagova, mada oni sami ne postaju deo finalnih struktura. Do stupnja sklapanja strukturnih komponenti u glavu i rep, replikacija DNK je dostigla svoju maksimalnu brzinu. Genomi se zatim umeću u prazne proteinske glave, repovi se dodaju, i ćelija domaćina se lizira da bi se omogućilo oslobađanje novih virusnih čestica.
Organizacija fagne genetičke mape često odražava sekvencu litičkog razvoja. Koncept operona je donekle doveden do ekstrema, u kome su geni koji kodiraju proteine sa srodnim funkcijama grupisani da bi se omogućila njihova kontrola sa maksimalnom efikasnošću. To omogućava kontrolu puta litičkog razvoja sa malim brojem regulatornih prekidača. Litički ciklus je pod pozitivnom kontrolom, tako da se svaka grupa gena faga može izraziti samo nakon davanja odgovarajućeg signala. Regulatorni geni funkcionišu u kaskadi u kojoj je gen izražen u jednom stupnju neophodan za sintezu gena koji su izraženi u sledećem stupnju. Tako je u svakom stupnju ekspresije jedan ili više aktivnih gena regulator koji je potreban u sledećem stupnju. Regulator može da ima oblik nove RNK polimeraze, sigma faktora koji preusmerava specifičnost domaćinove RNK polimeraze, ili antiterminacioni faktor koji omogućava čitanje nove grupe gena.
Prvi stupanj genske ekspresije se oslanja na transkripcioni aparat ćelije domaćina. Obično je samo nekoliko gena izraženo u tom stupnju. Njihovi promoteri su kao i promoteri gena domaćina. Ime ove klase gena zavisi od faga. U većini slučajeva, oni su poznati kao rani geni. Jedan od tih gena uvek kodira protein koji je neophodan za transkripciju sledeće klase gena. Druga klasa gena je poznata kao odloženi rana ili srednja grupa. Njeno izražavanje tipično počinje neposredno nakon kodiranja regulatornog proteina. U zavisnosti od prirode kontrolnog ciklusa, inicijalni set ranih gena može, ili ne mora da nastavi sa daljom ekspresijom. Često dolazi do redukcije izražavanja gena domaćina. Dva seta ranih gena obuhvataju sve funkcije faga izuzev formiranja proteinskog omotača i liziranja ćelije. Kad replikacija fagne DNK započne, vreme da se izraze kasni geni. Njihova transkripcija u ovom kasnom stupnju obično je uzrokovana regulatornim proteinom koje formiran u prethodnoj fazi. U slučaju lambda faga to je anterminacioni faktor, mada može da bude sigma faktor (kao kod SPO1).
Interpretacija genetičkog koda
Sekvenca kodirajućeg lanca DNK, koja se čita u smeru od 5' da 3', se sastoji od nukleotidnih tripleta (kodona) i odgovara aminokiselinskoj sekvenci proteina od N do C-terminusa. Sekvenciranje DNK i proteina omogućava direktno poređenje sekvenci nukleotida i aminokiselina. Transportna RNK je adapter koji povezuje kodon sa odgovarajućom aminokiselinom. Transportna TRK ima pivotalnu ulogu u sintezi proteina. Ona je posrednik koji omogućava translaciju kodona u aminokiseline. Značenje kodona koji predstavlja aminokiselinu je određeno njegovom transportnom RNK, dok značenje terminirajućih kodona proizilazi direktno iz proteinskih faktora.
Rešavanje genetičkog koda je originalno pokazalo da se genetička informacija održava u obliku nukleotidnih tripleta, ali nije bilo poznato kako kodoni specificiraju odgovarajuće aminokiseline. Pre razvoja sekvenciranja, relacije između aminokiseline i kodona su određene koristeći dva tipa in vitro studija. Sistem za translaciju sintetičkih polinukleotida je uveden 1961. i pokazano je da poliuridinska kiselina (poli(U)) sadrži instrukcije za formiranje polifenilalanina iz fenilalanina.[370] Drugi sistem je kasnije uveden u kome su trinukleotidi korišteni za oponašanje kodona uzrokujući vezivanje odgovarajuće tRNK za ribozom. Primenom kombinacije ove dve tehnike su određena značenja kodona.
Pošto postoji više kodona (61) nego amino kiselina (20), skoro sve aminokiseline su predstavljene sa više kodona. Jedini izuzeci su metionin i triptofan. Kodoni sa istim značenjem se nazivaju sinonimi. Pošto se genetički kod zapravo čita na iRNK, on se obično opisuje koristeći RNK baze: U, C, A i G. Kodoni koji predstavljaju istu ili srodne aminokiseline teže da imaju slične sekvence. Često baza u trećoj poziciji kodona nije značajna, jer četiri kodona koja se razlikuju u samo u trećoj bazi predstavljaju istu aminokiselinu. U nekim slučajevima razlika se pravi samo između purina i pirimidina u toj poziciji. Umanjena specifičnost na zadnjoj poziciji kodona je poznata kao degeneracija treće baze.
Interpretacija kodona zahteva sparivanje baza sa antikodonom odgovarajuće aminoacil-tRNK. Ova reakcija se odvije unutar ribozoma. Komplementarni trinukleotidi u izolaciji su suviše kratki da formiraju stabilne interakcije. Unutar ribozoma reakcija je stabilizovana njegovim aktivnom mestom. Formiranje baznih parova između kodona i antikodona nije samo pitanje sparivanja baza, A-U i G-C. Ribozom kontroliše sredinu na takav način da do konvencionalnog sparivanja dolazi na prve dve pozicije kodona, dok su dodatne reakcije moguće na trećoj bazi. Rezultat toga je da jedan aminoacil-tRNK molekul može da prepozna više od jednog kodona u skladu sa obrascem degeneracije. Na interakcije sparivanja može da utiče uvođenje specijalnih baza na iRNK. Modifikacije u ili blizo antikodona direktno utiču na sposobnost iRNK da formira interakcije sa željenim kodonima, dok modifikacije na drugim mestima mogu da utiču na ostale funkcije iRNK molekula.
Trend da su slične aminokiseline zastupljene srodnim kodonima umanjuje uticaj mutacija, i uvećava verovatnoću da nasumična promena jedne baze neće proizvesti promenu aminokiseline, ili bar ne promenu u kiselu različitog karaktera. Na primer, mutacija CUC u CUG nema efekta pošto oba kodona predstavljaju leucin, dok mutacija CUU u AUU proizvodi zamenu leucina sa izoleucinom, koji je blisko srodan.
- Tabela RNK kodona
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Tri kodona (UAA, UAG i UGA) koja ne predstavljaju aminokiseline koriste se za označavanje tačke završetka sinteze proteina. Kraj svakog gena je označen jednim od ovih kodona.
Poređenje DNK sekvenci sa odgovarajućim proteinskim sekvencama je pokazalo da se identičan set kodona koristi kod bakterija i eukariota. Konsekventno iRNK iz jedne vrste se obično može korektno translirati in vitro i in vivo protein sintetičkim aparatom druge vrste. Drugim rečima kodoni korišteni u iRNK jedne vrste imaju isto značenje za ribozome i tRNK drugih vrsta.
Univerzalnost koda ide u prilog pretpostavci da je on morao biti formiran veoma rano u toku evolucije. Originalno je možda postojao stereohemijski odnos između aminokiselina i kodona, koji je vremenom evoluirao do današnjeg sistema selekcijom osobina kao što su veća efikasnost i preciznost. Moguće je da je kod započet u primitivnim formama kod kojih je mali broj kodona korišten za predstavljanje malog broja aminokiselina, ili da je čak jedan kodon odgovarao bilo kom članu grupe aminokiselina. Preciznija značenja kodona i dodatne aminokiseline su možda evoluirali kasnije. Jedna mogućnost je da su u početku samo dve od tri baze korišćene, i da je diskriminacija treće pozicije razvijena kasnije. Do zamrzavanja dalje evolucije koda je moglo da dođe kad je sistem postao dovoljno kompleksan da bi dalje promene značenja kodona unele znatne poremećaje u postojeće proteine putem neprihvatljivih zamena aminokiselina. Njegova univerzalnost sugeriše da je do toga moralo doći u veoma ranim stupnjevima života tako da su ga svi živi organizmi nasledili iz iste grupe ćelija.
Varijacije standardnog genetičkog koda
Dok je postojanje malih varijacija standardnog koda bilo ranije predviđeno[373], takvi kodovi su otkriveni tek 1979. kad je pokazano da ljudski mitohondrijalni geni koriste alternativni kod. Mnoge male varijacije su otkrivene od tada[374], uključujući razne alternativne mitohondrijske kodove[375] i male varijante kao što je translacija kodona UGA kao triptofan u vrstama mikoplazme i translacija CUG kao serin umesto leucina kod nekih vrsta roda kandida.[376][377] Kod bakterija i arheja, GUG i UUG su često start kodoni, mada u retkim slučajevima, pojedini proteini mogu da koriste alternativne start kodone koji se normalno ne koriste kod tih vrsta.[374]
U nekim proteinima, nestandardne aminokiseline su zamenjene za standardne stop kodone u zavisnosti od signalne sekvence informacione RNK. Na primer, UGA može da kodira selenocistein, i UAG može da kodira pirolizin. Selenocistein se smatra dvadeset prvom aminokiselinom, i pirolizin se smatra dvadest drugom.[374] Uprkos tih razlika, svi poznati prirodni kodovi su veoma slični jedan drugom, i mehanizam kodiranja je sličan za sve organizme: trobazi kodoni, tRNK, ribozomi, čitanje koda u istom smeru i translacija koda kodon po kodon u sekvencu aminokiselina. Problem poremećaja uzrokovanih promenama značenja kodona je manje ozbiljan kod mitohondrija, jer njihov DNK kodira mali broj proteina (~10). Promenjeni kodoni verovatno nisu bili ekstenzivno korišćeni u lokacijama gde bi supstitucije aminokiselina bile štetne. Raznovrsne promene nađene u mitohondrijama različitih vrsta sugerišu zasebnu evoluciju, sa odsustvom nasleđivanja od zajedničkog drevnog mitohondrijskog koda.
Minimum od 31 tRNK molekula (ne računajući inicijator) je neophodan za prepoznavanje 61 kodona (najmanje dva tRNK molekula su neohodna za svaku kodonsku familiju i jedan je neophodan po kodonskom paru). Kod sisarskih mitohondrija postoje samo 22 različita tRNK molekula. Kritična osobina je pojednostavljenje kodon-antikodon uparivanja tako da jedan tRNK prepoznaje sva četiri člana kodonske familije. Time se minimalni broj tRNK molekula neophodnih za prepoznavanje svih uobičajenih kodona redukuje do 23. Upotreba AGA(G) za terminaciju redukuje ovaj broj dalje do 22. Kod svih osam kodonskih familija, sekvenca tRNK kodona sadrži nepromenjen U na prvoj poziciji antikodona. Preostali kodoni su grupisani u parove u kojima se svi kodoni koji se završavaju pirimidinima čitaju modifikovanim G u antikodonu, i svi kodoni koji se završavaju purinima se čitaju modifikovanim U antikodona, kao što je predviđeno hipotezom o neodređenosti trećeg nukleotida kodona. Komplikacija sa UGG kodonom je izbegnuta promenom koda da čita UGA sa UGG kao triptofan, i kod sisara AUA prestaje da predstavlja izoleucin i umesto njega se čita kao i AUG za metionin. Time se omogućava čitanje svih kodona izvan kodonskih familija sa 14 parova. Skup od 22 identifikovana gena stoga kodiraju 14 tRNK molekula koji predstavljaju parove, i 8 tRNK molekula koji predstavljaju familije. To ostavlja dva nekorištena terminaciona kodona UAG i UAA koje tRNK molekuli ne prepoznaju, zajedno sa kodonskim parom AGA(G). Slična pravila slede mitohondrije gljivica.
Pored opštih promena koda, postoje specifične promene u čitanju individualnih gena. Specifičnost takvih promena podrazumeva da je čitanje pojedinog kodona zavisno od okružujućih baza. Upadljiv promer je inkorporacija modifikovane amino kiseline selenocisteina na pojedinim UGA kodonima unutar gena koji kodiraju selenoproteine kod prokariota i eukariota. Obično ti proteini katalizuju reakcije oksido-reduciju, i sadrže jedan selenocisteinski ostatak, koji formira deo aktivnog mesta. Najbolje je poznata upotreba UGA kodona u tri E. coli gena koji kodiraju format dehidrogenazne izozime. Interni UGA kodon se čita pomoću seleno-Cys-tRNK. Ova neobična reakcija je određena lokalnom sekundarnom strukturom iRNK, posebno prisustvom matične petlje u sekvenci neposredno ispod UGA. Mutacije u četiri Sel gena kreiraju deficit selenoproteinske sinteze.
Funkcija tRNK u proteinskoj sintezi je izvršena nakon prepoznavanja kodona unutar ribozoma. Interakcija između antikodona i kodona se sastoji od sparivanja baza na način koji ide izvan uobičajenog formiranja G-C i A-U veza. Pravila kojima se pokoravaju ove interakcije se mogu izvesti iz sekvenci antikodona. Sposobnost bilo koje tRNK da odgovori na određeni kodon se može direktno meriti putem testa trinukleidnog vezivanja ili njegovom upotrebom u in vitro proteinskom sintetičkom sistemu. Sam genetički kod daje važne indikacije o procesu prepoznavanja kodona. Obrazac degeneracije treće baze ukazuje da je u skoro svim slučajevima ta baza ili nije relevantna, ili se razlika pravi samo između purina i pirimidina. Postoji osam familija kodona u kojima sva četiri kodona imaju zajedničke prve dve baze, tako da treće nema ulogu u specificiranju aminokiseline. Kod sedam kodonskih parova značenje ne zavisi od tipa pirimidina u trećoj poziciji, i kod pet kodonskih parova to je slučaj sa purinom.
Ako se na genetički kod gleda u suprotnom smeru, postoje samo tri slučaja kod kojih se jedinstveno značenje dobija prisustvom određene baze u trećoj poziciji: AUG (za metionin), UGG (za triptofan), i UGA (terminacija). Iz toga sledi da C i U nemaju jedinstveno značenje u trećoj poziciji, dok A nikad ne označava jedinstvenu aminokiselinu. Pošto je antikodon komplementaran sa kodonom, prva baza antikodona se sparuje sa trećom bazom kodona. Na primer kodon ACG / antikodon CGU označava:
Kodon 5' A C G 3' Antikodon 3' U G C 5'
| Prva baza antikodona |
Treća baza kodona |
|---|---|
| U | A ili G |
| C | samo G |
| A | samo U |
| G | C ili U |
Često jedna iRNK može da prepozna više kodona. To znači da baze u prvoj poziciji antikodona moraju da imaju sposobnost formiranja interakcija sa alternativnim bazama u odgovarajućoj trećoj poziciji kodona. Sparivanje baza u toj poziciji ne može da bude ograničeno na uobičajene G-C i A-U parove. Pravila koja određuju obrasce prepoznavanja se mogu sumirati hipotezom fleksibilnosti, koja navodi da sparivanje između kodona i antikodona u prve dve pozicije kodona uvek sledi uobičajena pravila, dok se fleksibilnost javlja u trećoj poziciji. Do toga dolazi zato što konformacija petlje tRNK antikodona omogućava fleksibilnost na prvoj bazi antikodona. Pravila prepoznavanja treće baze kodona dozvoljavaju dodatno sparivanje između G i U. Ta jedna promena formira obrazac baznog sparivanja u kome A više nema jedinstveno značenje u kodonu, jer U koje ga prepoznaje mora isto tako da prepozna G. Slično tome C takođe više nema jedinstveno značenje, jer G koje ga prepoznaje mora takođe da prepozna U. Prepoznavanje jedinstvenih kodona je moguće samo kad je treća baza G ili U. Ta opcija se ne koristi često. UGG i AUG su jedini primeri prvog tipa, a nema primera drugog tipa.
G-U parovi su česti u RNK dupleks strukturama. S druge strane formiranje stabilnih kontakta između kodona i antikodona, gde se samo tri para baza mogu formirati je znatno ograničenije, i stoga G-U parovi mogu da doprinesu samo u zadnjoj poziciji kodona.
Uticaj modifikovanih baza na tRNK
Transportna RNK je jedinstvena među nukleinskim kiselinama po svom sadržaju „neuobičajenih“ baza. Neuobičajena baza je svaki purinski ili pirimidinski prsten izuzev uobičajenih A, G, C i U koje ulaze u sastav svih RNK. Sve druge baze se formiraju putem modifikacije jedne od četiri baze nakon njene inkorporacije u poliribonukleinski lanac. Sve klase RNK pokazuju izvestan stupanj modifikacija, ali svim slučajevima izuzev tRNK to je ograničeno na veoma jednostavne slučajeve, kao što je adicija metil grupa. Kod tRNK se javlja širok niz modifikacija, koje se kreću od od jednostavne metilacije do potpune rekonstrukcije purinskog prstena. Modifikacije se javljaju u svim delovima tRNK molekula. Lista modifikovanih nukleotida sadrži oko pedeset baza.
Modifikacije pirimidina (C i U) su manje kompleksne od modifikacija purina (A i G). Osim modifikacija samih baza, dolazi i do metilacije na 2'-O poziciji riboznog prstena. Najčešće modifikacije uridina su jednostavne. Metilacija u poziciji 5 kreira ribotimidin (T). Baza je ista sa bazom timidina, ali je vezana za ribozu umesto dezoksiriboze. Dihidrouridin (D) se formira zasićenjem dvostruke veze, čime se menja prsten. U pseudouridinu (Ψ) su zamenjene pozicije N i C atoma prstena, dok 4-tiouridin ima sumpor umesto kiseonika. Nukleozid inozin se normalno javlja u ćelijama kao intermedijar puta biosinteze purina. On se ne unosi direktno u RNK, nego se formira modifikacijom adenozina. Druge modifikacije anadenozina su dodaci kompleksnih grupa. Dve kompleksne serije nukleozida se formiraju promenom guanozina. Q baze poput kjuozina, imaju dodatni pentenilni prsten dodat preko NH veze na metil grupu 7-metilguanozina. Pentenilni prsten može da nosi različite grupe. Y baze poput viozina, imaju dodatni prsten kondenzovan sa purinskim prstenom. Taj dodatni prste ima dugačak lanac na kome mogu da budu prisutne razne grupe.[1]
Reakcija modifikacija se obično sastoje od promene ili adicije postojećih baza u tRNK. Izuzetak je sinteza Q baza, gde specijalni enzimi zamenjuju guanozinski ostatak kjuozinskim ostatkom u RNK. Reakcija se sastoji od raskidanja i formiranja veza sa obe strane nukleozida. Ostali modifikovani nukleozidi se sintetišu specifičnim tRNK modifikujućim enzimima. Originalni nukleozid prisutan u datoj poziciji se može odrediti bilo poređenjem sekvence tRNK sa genom ili putem izolovanja prekursorskog molekula kome nedostaju neke ili sve modifikacije. Sekvence prekursora pokazuju da se različite modifikacije uvode u različitim stupnjevima tokom maturacije DNK. Neke modifikacije su konstantne osobine svih tRNK molekula, na primer D ostaci po kojima je formirano ime D ruka, i Ψ nađen u TΨC sekvenci. Na 3' kraju antikodona se uvek javlja modifikovani purin, mada modifikacija može znatno da varira. Druge promene su specifične za pojedine tRNK molekule ili grupe tRNK molekula. Na primer, viozinske baze su karakteristika tRNKPhe kod bakterija, kvasaca i sisara.
Osobine koje prepoznaju tRNK modifikujući enzimi su nepoznate. Kad je specifična modifikacija prisutna na više mesta u tRNK molekulu to ne mora da znači da je isti enzim napravio sve promene, na primer, različiti enzimi mogu da budu potrebni za sintezu pseudouridina na svakoj lokaciji. Postoji znatan broj modifikujućih enzima. Neki enzimi vrše pojedinačne reakcije sa individualnim RNK molekulima, dok drugi imaju širi opseg supstrata. Pojedine promene se vrše putem sukcesivnog dejstva više enzima. Najdirektniji učinak modifikacije je u antikodonu, gde promena sekvence utiče na sposobnost vezivanja kodona, te određuje značenje tRNK molekula. Promene na drugim mestima u blizini antikodona isto tako utiču na vezivanje kodona. Promena baza antikodona može da formira dodatne obrasce uparivanja baza, kao što su tiouracil-guanin, uracil-inozin, i adenin-inozin.
Interakcije sa proteinima
Funkcionisanje DNK je zavisno od interakcija sa proteinima. Proteinske interakcije mogu da budu nespecifične, ili se protein može vezati za specifičnu DNK sekvencu. Enzimi se takođe vezuju za DNK, i među njima su posebno važne polimeraze koje kopiraju DNK sekvencu tokom transkripcije i replikacije DNK.
DNK vezujući proteini

|

Strukturni proteini koji se vezuju za DNK su dobro izučeni primeri nespecifičnih DNK-protein interakcija. U hromozomima, DNK je smeštena u kompleksima sa strukturnim proteinima. Ti proteini organizuju DNK u kompaktne hromatinske strukture. Kod eukariota u njima je DNK vezana u komplekse sa histonima, dok je kod prokariota više tipova proteina prisutno.[379][380] Histoni formiraju komplekse u obliku diska, nukleozome. Te nespecifične interakcije se formiraju putem jonskih veza između baznih ostataka histona sa kiselom šećerno fosfatnom osnovom DNK, te su stoga u znatnoj meri nezavisne od sekvence baza.[48]
Hemijske modifikacije baznih aminokiselinskih ostataka su metilacija, fosforilacija i acetilacija.[381] Te hemijske promene menjaju jačinu interakcije između DNK i histona, što čini DNK manje dostupnom za transkripcione faktore te se menja brzina transkripcije.[382] Drugi nespecifični DNK vezujući proteini u hromatinu su grupe proteina visoke mobilnosti, koje se vezuju da bi se savila ili iskrivila DNK.[383] Ti proteini su važni u savijanju grupa nukleozome i njihovog organizovanja u veće strukture koje sačinjavaju hromozome.[384]
Posebna grupa DNK-vezujućih proteina su proteini koji se specifično vezuju jednolančanu DNK. Kod ljudi, replikacioni protein A je najbolje izučeni član ove familije. On učestvuje u procesima gde je dvostruki heliks razdvojen, uključujući replikaciju DNK, rekombinaciju i popravku DNK.[385] Vezivanje tih proteina stabilizuje jednolančanu DNK i štiti je od formiranja matičnih petlji (uzrokovanih uparivanjem baza isto lanca), kao i od degradacije nukleazama.
Drugi proteini su evoluirali da se vežu za specifične DNK sekvence. Među njima su najintenzivnije studirani različiti transkripcioni faktori, koji regulišu transkripciju. Svaki transkripcioni faktor se vezuje za jedan specifičan segment DNK sekvence, i aktivira ili inhibira transkripciju gena koji imaju te sekvence u blizini njihovih promotera. Transkripcioni faktori deluju na dva načina. Oni mogu da se vežu za RNK polimerazu odgovornu za transkripciju, bilo direktno ili putem drugih posredničkih proteina. Time se dovodi polimeraza na promoter i omogućava početak transkripcije.[386] Alternativno, transkripcioni faktori se mogu vezati za enzime koji modifikuju histone na promoterima. Time se menja pristupačnost DNK templata za polimeraze.[387]
Ovi DNK ciljevi se mogu javiti širom genoma jednog organizma, te stoga promene u aktivnosti jednog tipa faktora transkripcije faktora mogu da utiču na hiljade gena.[388] Konsekventno, ti proteini su često meta za procese prenosa signala koji kontrolišu odgovore na promene u okolini, ili na ćelijsku diferencijaciju i razvoj. Specifičnost interakcija transkripcionih faktora sa DNK molekulom se proizvodi kontaktom proteina sa ivicama DNK baza, što im omogućava da „čitaju“ DNK sekvence. Većina tih interakcija sa bazama se odvija u glavnom žlebu, gde su baze najpristupačnije.[22]
Enzimi koji modifikuju DNK
Nukleaze


Nukleaze su enzimi koji presecaju DNK lance putem katalizovanja hidrolize fosfodiestarskih veza. Nukleaze koje hidrolizuju nukleotide sa krajeva DNK lanaca se nazivaju eksonukleaze, dok endonukleaze mogu da presecaju unutar lanaca.
Egzonukleaze odvajaju jedan po jedan nukleotid sa kraja polinukleotidnog lanca. Reakcijom hidrolize se raskidaju fosfodiesterske veze sa bilo 3’ ili 5’ kraja. Eukarioti i prokarioti imaju tri tipa egzonukleaza koje učestvuju u normalnom prometu iRNK: 5’ do 3’ egzonukleaza, koja je zavisna od dekapirajućeg proteina, 3’ do 5’ egzonukleaza, koja je nezavistan protein, i poli(A)-specifična 3’ do 5’ egzonukleaza.[393][394]
Najčešće korišćene nukleaze u molekularnoj biologiji su restrikcione endonukleaze, koje presecaju DNK na specifičnim sekvencama. Na primer, EcoRV enzim prepoznaje šestobaznu sekvencu 5′-GAT|ATC-3′ i preseca je na mestu označenom vertikalnom linijom. U prirodi, ti enzimi štite bakterije protiv fagne infekcije. Nakon unosa fagne DNK u bakterijsku ćeliju na nju deluje restrikcioni modifikacioni sistem.[395] U tehnologiji se nukleaze specifične za pojedine sekvence koriste u molekulskom kloniranju i DNA rekombinaciji.
Flap endonukleaze (takođe poznate kao 5' nukleaze u starijoj literaturi) su klasa nukleolitičkih enzima koji deluju kao 5'-3' egzonukleaze i strukturno specifične endonukleaze na specijalizovanim DNK strukturama koje se javljaju tokom replikacije, popravke i rekombinacije DNK. Flap endonukleaze su identifikovane kod eukariota, prokariota, arheja i nekih virusa. Organizmi mogu da imaju više od jednog homologa. Ova redundantnost može da služi kao indikacija važnosti ovih enzima. Kod prokariota, flap endokrinaze su prisutne na N-terminusnom domenu DNK polimeraze I. Neki prokarioti kodiraju i drugi homolog.[396][397][398]
| Enzim | Izvor | Sekvenca prepoznavanja | Presecanje |
|---|---|---|---|
| EcoRI | Ešerihija koli | 3'CTTAAG |
3'---CTTAA G---5' |
Mnoge endonukleaze presecaju DNK u pozicijama koje nisu direktno jedna naspram druge, te se staraju prepusti. Primer takve nukleaze je EcoRI.[399][400] Kad ovaj enzim naiđe na svoju sekvencu prepoznavanja, on preseca svaki lanac između G i najbližeg A baznog ostatka. Nakon presecanja, novonastali fragmenti se drže zajedno samo posredstvom relativno slabih vodoničnih veza koje sprežu komplementarne baze. Mala jačina ovih veza omogućava DNK fragmentima da se odvoje jedan od drugog. Svaki rezultujući fragment ima istureni 5' kraj koji se sastoji od nesparenih baza. Drugi enzimi presecaju DNK lance na suprotan način tako da se formiraju istureni 3' krajevi. Ovi jednolančani krajevi, 3' i 5', se ponekad nazivaju lepljivi krajevi zato što oni imaju tendenciju da se vezuju sa komplementarnim sekvencama baza. Drugim rečima, ako nespareni segment baza (5' A A T T 3') naiđe na drugi nespareni segment sa sekvencom (3' T T A A 5') oni će se vezati jedan za drugi. Ligaze zatim spajaju fosfatne osnove ta dva molekula. Ćelijsko poreklo lepljivih krajeva nema uticaja na njihovu sposobnost spajanja. Svaki par komplementarnih sekvenci ima tendenciju vezivanja, čak i u slučaju kombinacija dugačkih humanih DNK sekvenci i bakterijske DNK. Ovo svojstvo omogućava proizvodnju rekombinantnih DNK molekula, koji se sastoje od DNK fragmenata iz različitih izvora, i koji su formirani primenom tehnika genetičkog inženjeringa.
Ligaze
Enzimi DNK ligaze mogu da spoje odsečene ili pokidane DNK lance.[401][402][403] Ligaze su posebne važnosti u replikaciji DNK zaostajućeg lanca, gde one spajaju kratke segmente DNK proizvedene na replikacionoj viljušci u kompletnu kopiju DNK templeta. One se takođe koriste u DNK popravci i genetičkoj rekombinaciji.[401]
Mehanizam rada DNK ligaze se sastoji od formiranja dve kovalentne fosfodiestarske veze između 3' hidroksilnog kraja jednog nukleotida, (akceptora) i 5' fosfatnog kraja drugog (donora). ATP je neophodan za odvijanje reakcije posredovane ligazom. Reakcija se odvija u tri stepena: (1) adenilacija (adicija AMP-a) aminokiselinskog ostatka u aktivnom centru enzima, pirofosfat se oslobađa; (2) transfer AMP-a na 5' fosfat donora, čime se formira pirofosfatna veza; (3) formiranje fosfodiestarske veze između 5' fosfata donora i 3' hidroksila akceptora.[404][405][406]
Ligaza takođe može da spaja tupe krajeve, mada su više koncentracije enzima i različiti eksperimentalni uslovi neophodni.[407][408]
Kod sisara postoje četiri specifična tipa ligaze.[409]
DNA ligaza kod E. coli koristi energiju stečenu raskidanjem nikotinamid adenin dinukleotida (NAD) da formira fosfodiestersku vezu.[357] DNK ligaza eukariota kao i nekih mikroba koristi adenozin trifosfat (ATP) umesto NAD-a.[357]
Topoizomeraze

Postoje tri glavna tipa topologije: supernamotavanje, učvoravanje i katenacija. Izvan esencijalnih procesa replikacije ili transkripcije, DNK se održavana u kompaktnom obliku, i ova tri topološka stanja pomažu u tome.[58] Međutim, u toku transkripcije ili replikacije DNK mora biti slobodna, te ova tri stanja znatno ometaju proces. Dodatno, tokom replikacije, novoformirani i originalni DNK dupleksi se isprepleću i moraju biti kompletno razdvojeni da bi se održao genomski integritet ćelija tokom deobe. Kako transkripcioni mehur napreduje, DNK ispred traskripcione viljuške postaje prekomerno uvijena, ili pozitivno supernamotana, dok DNK iza transkripcionog mehura postaje odvijena, ili negativno supernamotana.
Topoizomeraze mogu da reše ove topološke probleme. One su enzimi sa nukleaznom i ligaznom aktivnošću. Ovi proteini menjaju količinu supernamotavanja u DNK molekulu. Deo enzima ove grupe deluje tako što presecaju DNK heliks i omogućavaju jednoj sekciji da rotira, čime se umanjuje njen nivo supernamotavanja. Enzim zatim zatvara DNK prekid.[57] Drugi tipovi ovih enzima imaju sposobnost prekidanja jednog DNK heliksa i zatim propuštanja drugog lanca kroz ovaj prekid, pre nego što ponovo spoje heliks.[411]
Topoizomeraze se dele na dva tipa na osnovu broja presečenih lanaca u jednom krugu delovanja:[412] Obe klase enzima koriste konzervirani tirozin, međutim ovi enzimi su strukturno i mehanistički različiti.
- Tip I topoizomeraza preseca jedan lanac DNK dvostrukog heliksa, dolazi do relaksacije, i nakon toga presečeni lanac se poveže. Tip I topoizomeraze se dalje dele u dve podklase: tip IA topoizomeraze, koje imaju zajedničke mnoge strukturne i mehanističke osobine sa topoizomerazama tipa II, i tip IB topoizomeraze, koje koriste kontrolisani rotacioni mehanizam. Primeri tip IA topoizomeraza su topo I i topo III. Istorijski, tip IB topoizomeraze su nazivane eukariotske topo I, mada su IB topoizomeraze prisutne u svim životnim domenima. Tip IA topoizomeraze formiraju kovalentni intermedijar sa 5' DNK krajem, dok IB topoizomeraze formiraju kovalentni intermedijar sa 3' DNA krajem. Nedavno je tip IC topoizomeraza bila identifikovana. Ona se naziva topo V. Dok je ona strukturno jedinstvena u odnosu na tip IA i IB topoizomeraze, ona ima sličan mehanizam sa topoizomerazom tipa IB .
- Tip II topoizomeraza preseca oba lanca DNK dvostrukog heliksa.[410] Takođe postoje dve podklase: tip IIA i tip IIB topoizomeraze, koje imaju slične strukture i mehanizam. Primeri tip IIA topoizomeraze su eukariotski topo II, E. coli giraza, i E. coli topo IV. Primer tipa IIB topoizomeraze je topo VI.
Oba tipa (I i II) topoizomeraze menjaju DNK koeficijent vezivanja.[413] Topoizomeraze tipa IA menjaju koeficijent vezivanja za jedan, tip IB i tip IC topoizomeraze menjaju taj parametar za bilo koji ceo broj, dok tip IIA i tip IIB topoizomeraze menjaju koeficijent vezivanja za dva.
Helikaze
Helikaze su proteini koji su tip molekulskih motora. One koriste hemijsku energiju nukleozid trifosfata, predominantno ATP-a, da raskinu vodonične veze između baza i da odviju DNK dvostruki heliks u jednostruke lance.[414] Ovi enzimi su esencijalni za većinu procesa u kojima je neophodno da se razdvoje lanci DNK lanci (replikacija DNK, transkripcija, translacija, rekombinacija, DNK popravka, biogeneza ribozoma). One se postepeno kreću duž dvostrukog lanca nukleinske kiseline. Pravac i mehanizam dejstva su zavisni od tipa enzima.[415]

Helikaze poprimaju različite strukture i oligomerizaciona stanja. DnaB helikaze imaju oblik heksamernog prstena. Drugi enzimi su aktivni u obliku monomera ili dimera. Ispitivanja su pokazala da helikaze mogu da deluju pasivno, čekajući da dođe do nekatalisanog odvijanja nakon čega one razdvajaju lance,[417] ili da aktivno učestvuju u separaciji lanaca koristeći energiju generisanu hidrolizom ATP-a.[418] U ovom drugom slučaju, helikaze deluju poput molekulskih motora, odvijajući i translocirajući svoj supstrat koristeći hemijsku energiju.[419] Helikaze mogu da funkcionišu mnogo brže in vivo nego in vitro usled prisustva pomoćnih proteina koji posreduju destabilizaciju DNK račve.[419]
Helikaze se klasifikuju u nekoliko superfamilija. Sve helikaze vezuju ATP, i stoga sadrže klasične motive: Valker A (petlja vezivanja fosfata ili P-petlja) i Valker B (aspartična kiselina za vezivanje Mg2+).[415]
- Superfamilija I: UvrD (DNK popravka kod E. coli), Rep (DNK replikacija kod E. coli), PcrA (rekombinacija kod Staphylococcus aureus), Dda (inicijacija replikacije bakteriofaga T4), RecD (rekombinaciona popravka kod E. coli), TraI (konjugativni DNK transfer F-plazmida). Ova familija obuhvata RNK helikaze koji učestvuju u odmotavanju dupleksa tokom viralne RNK replikacije.[420]
- Superfamilija II: RecQ (DNK popravka E. coli), eIF4A (RNK translacija kod pekarskog kvasca), WRN (DNK popravka), NS3[421] (replikacija Hepatitis C virusa)
- Superfamilija III: LTag (replikacija SV40 virusa), E1 (replikacija Ljudskog papilomskog virusa), Rep (replikacija, viralna integracija, i pakovanje Adeno asociranog virusa). Superfamilija 3 se sastoji od helikaza koje uglavnom kodiraju mali DNK virusi i neki od velikih nukleocitoplazmatičnih DNK virusa.[50][422]
- Familija slična DnaB helikazi: DnaB (replikacija E. coli), gp41 (DNK replikacija bakteriofaga T4), T7gp4 (DNK replikacija bakteriofaga T7).
- Familija slična Ro faktoru: Ro (terminacija transkripcije E. coli).
Ove superfamilije ne obuhvataju sve poznate helikaze. Na primer, XPB[423] i ERCC2[424] su helikaze koje nisu svrstane u gore navedene familije.
Defekti gena koji kodiraju helikaze mogu da uzrokuju Vernerov sindrom. To je poremećaj koji je prepoznatljiv po pojavi prevremenog starenja.[425][426]
Polimeraze

Polimeraze su enzimi koji sintetišu polinukleotidne lance iz nukleozidnog trifosfata. Sekvence njihovih produkata su kopije postojećih polinukleotidnih lanaca – koji se nazivaju templetima. Ovi enzimi funkcionišu tako što dodaju nukleotide na 3′ hidroksilnu grupu prethodnog nukleotida u DNK lancu. Konsekventno, sve polimeraze idu u 5′ ka 3′ smeru.[428] U aktivnom mestu tih enzima, novi nukleozid trifosfat formira bazni par sa templetom, čime omogućava polimerazi da precizno sintetiše komplementarni lanac. Polimeraze se klasifikuju po tipu templeta koji koriste.
Tokom replikacije DNK, DNK polimeraze formiraju kopiju DNK sekvence. Preciznost je od vitalnog značaja u ovom procesu, tako da mnoge polimeraze imaju sposobnost vršenja korekcija. Polimeraza lako prepoznaje povremene greške u reakciji sinteze zato nema baznog sparivanja između neusklađenih nukleotida. Ako se neslaganje detektuje, 3′ ka 5′ egzonukleaza se aktivira i pogrešna baza se uklanja.[429] U većini organizama, DNK polimeraze funkcionišu unutar velikih kompleksa. Oni se nazivaju replizomima i sadrže niz pomoćnih podjedinica, kao što su DNK stege i helikaze.[430]
DNK polimeraze koje su zavisne od RNK su specijalizovana klasa polimeraza koja kopira sekvencu RNK lanca u DNK. U ovu klasu se ubrajaju reverzna transkriptaza, koja je viralni enzim koji učestvuje u infekciji ćelija retrovirusima, i telomeraza, koja je neophodna za replikaciju telomera.[112][431] Telomeraza je neobična polimeraza zato što sadrži svoj sopstveni RNK templet, kao integralni deo svoje strukture.[114]
Transkripciju izvodi DNK-zavisna RNK polimeraza koja kopira sekvencu DNK lanca u RNK. Da bi započela transkripciju gena, RNK polimeraza se veže za DNK sekvencu promotera i razdvoji DNK lance. Ona zatim kopira sekvencu gena na iRNK transkript dok ne dođe do DNK regiona koji se naziva terminator, gde se zaustavlja i odvaja od DNK. Kao i DNK polimeraze, RNK polimeraza II, enzim koji transkribuje većinu gena u genomu eukariota, operiše kao deo većeg proteinskog kompleksa sa višestrukim regulatornim podjedinicama.[432][433][434] RNK polimeraza II je kompleks sa masom od 550 kDa, koji se sastoji od dvanaest podjedinica. RNAP II je najbolje ispitani tip RNK polimeraze. Znatan broj transkripcionih faktora je neophodan da bi se ovaj enzim vezao za promotere i počeo transkripciju.
Evolucija
DNK sadrži genetičke informacije koje omogućavaju svim životnim formama da funkcionišu, rastu i razmnožavaju se. Međutim, nije jasno koliko dugo je tokom zadnjih četiri milijarde godina istorije života DNK obavljala tu funkciju. Po jednoj pretpostavci najranije forme života su koristile RNK kao osnovu genetičkog materijala.[435][436] Moguće je da je RNK delovala kao centralni deo ranog ćelijskog metabolizma pošto ona ima sposobnost prenosa genetičke informacije, kao i izvođenja katalize u okviru ribozima.[437] Takav drevni RNK svet u kome bi nukleinska kiselina bila korišćena za katalizu i genetiku je možda uticao na evoluciju sadašnjeg genetičkog koda baziranog na četiri nukleotidne baze. Do toga bi došlo, pošto bi broj različitih baza u takvom organizmu bio kompromis između malog broja baza kojim se povećava replikaciona preciznost i većeg broja baza kojim se uvećava katalitička efikasnost ribozima.[438]
Ne postoje direktni dokazi za postojanje takvih drevnih genetičkih sistema. Razlog za to je DNK ekstrakcija iz velike većine fosila nije moguća pošto DNK može da opstane manje od milion godina. U vlažnoj sredini ona se vremenom razlaže u kratke fragmente.[439] Uprkos tome, nalazi starije DNK su bili objavljeni. Na primer, poznat je izveštaj o izolaciji održive bakterije iz kristala soli starog 250 miliona godina.[440] Ti nalazi su kontroverzni.[441][442]
Jedan izveštaj objavljen avgusta 2011. koji je baziran na NASA ispitivanjima meteorita nađenog na Zemlji sugeriše da gradivni blokovi DNK (adenin, guanin i srodni organski molekuli) možda imaju vanzemaljsko poreklo.[443][444][445]
Tehnološka primena
Genetički inženjering
Genetički inženjering je direktna ljudska manipulacija genoma nekog organizma koristeći modernu DNK tehnologiju. Ona obuhvata uvođenje strane DNK ili sintetičkih gena u organizam. Uvođenje nove DNK ne zahteva upotrebu klasičnih genetičkih metoda, međutim tradicionalni metodi uzgoja se tipično koriste za propagaciju rekombinantnih organizama. Organizam koji je formiran uvođenjem rekombinantne DNK se smatra genetički modifikovanim organizmom. Prvi genetički modifikovani organizmi su bile bakterije 1973. i zatim miševi 1974. Bakterije koje proizvode insulin su komercijalizovane 1982. i genetički modifikovana hrana je u prodaji od 1994.
Methode su razvijena za DNK prečišćavanje iz ćelija organizma, kao što je fenol-hloroformna ekstrakcija, i za njenu laboratorijsku manipulaciju, npr. restrikciona razlaganja i polimerazna lančana reakcija. Moderna biologija i biohemija intenzivno koriste te tehnike u okviru rekombinantne DNK tehnologije. Rekombinantna DNK je veštačka DNK sekvenca koja je formirana od drugih DNK sekvenci. Takve sekvence se mogu uneti u organizam u obliku plazmida ili u odgovarajućem formatu, koristeći viralni vektor.[446]
Najčešća forma genetičkog inženjeringa je umetanje novog genetičkog materijala na nepoznatoj lokaciji u genomu domaćina. To se ostvaruje izolovanjem i kopiranjem željenog genetičkog materijala koristeći metode molekulskog kloniranja da bi se formirala DNK sekvenca kaja sadrži neophodne genetičke elemente za ekspresiju, i zatim njeno umetanje u organizam domaćina. Druge forme genetičkog inženjeringa su ciljanje gena (zamena bazirana na homolognoj rekombinaciji) i uklanjanje (nokaut) specifičnih gena putem projektovanih nukleaza kao što su nukleaze cinkovog prsta,[447] ili projektovane homing endonukleaze.[448]
Tehnike genetičkog inženjeringa se koriste u brojnim poljima naučnih istraživanja, biotehnologiji, i medicini. Genetički modifikovani organizmi nalaze primenu u izradi proizvoda kao što su rekombinantni proteini, oni se koriste u medicinskim istraživanjima,[449] i kao poljoprivredni usevi.[450][451][452] Lekove kao što su insulin i ljudski faktor rasta proizvode bakterije, eksperimentalni miševi kao što su onkomiševi i nokaut miševi se koriste u istraživanjima, repelanti insekata i/ili usevi tolerantni na herbicide su komercijalizovani. Genetički modifikovane biljke i životinje koje imaju sposobnost proizvodnje biotehnoloških lekova uz manje troškove proizvodnje od sadašnjih metoda se takođe razvijaju. FDA je 2009. odobrila prodaju farmaceutskog proteina antitrombina proizvedenog u mleku genetički modifikovane koze.[453]
Forenzika
Forenzička analiza može da koristi DNK iz krvi, sperme, kože, pljuvačke ili kose nađene na mestu zločina za identifikaciju podudarajuće DNK neke osobe, kao što je počinilac. Ovaj proces se formalno naziva DNK profilisanje, a poznat je i kao „uzimanje genetičkog otiska“. Mada je 99,9% DNK sekvenci isto kod svih ljudi, dovoljna količina DNK se razlikuje, tako da moguće razlikovati jednu osobu od druge, ukoliko one nisu monozigotni blizanci.[454] U DNK profilisanju, dužina promenljivih sekcija ponavljajuće DNK, kao što su kratka tandemna ponavljanja i minisateliti,[455] se porede između ljudi. Taj metod je obično izuzetno pouzdana tehnika za identifikaciju podudarne DNK.[456] Međutim, identifikacija može da bude komplikovana ako je mesto zločina kontaminirano sa DNK-om od nekoliko ljudi.[457][458] DNK profilisanje je razvio 1984. britanski genetičar Sir Alek Džefriz,[459] i prvi put je korišćeno u forenzičkoj nauci da se osudi Kolin Pičfork 1988, u slučaju Enderbi ubistava.[460]
Razvoj forenzičke nauke, i sposobnosti da se dobiju genetička podudaranja korišćenjem veoma malih uzoraka krvi, kože, pljuvačke ili kose je dovela do ponovnog ispitivanja brojnih slučajeva. Genetičkim profilisanjem se može proizvesti evidencija koja nije postojala tokom originalnog ispitivanja. U kombinaciji sa uklanjanjem zakona o ponovljenim sudskim procesima, postalo je moguće da se ponovo otvore slučajevi sa ranijim neuspešnim presudama usled nedostatka evidencije. Od ljudi optuženih za teške zločine se zahteva da daju uzorak DNK radi profilisanja. Rezultati DNK profilisanja se često dovode u pitanje tvrdnjama o kontaminaciji tokom uzimanja uzoraka. To je dovelo do razvoja detaljnih i strogih procedura rukovanja uzorcima novih slučajeva teških krivičnih dela.[461] DNK profilisanje se takođe koristi za identifikaciju žrtava masovnih incidenata.[462] Osim pozitivne identifikacije tela ili delova tela ozbiljnih incidenata, DNK profilisanje se uspešno koristi i za identifikaciju žrtava u masovnim ratnim grobnicama, putem DNK poređenja sa članovima porodice.[463]
Bioinformatika
Bioinformatika se bavi manipulacijom, pretraživanjem i analizom bioloških podataka, što obuhvata DNK sekvence. Termin bioinformatika je skovan pre početka „genomske revolucije“. Paulien Hogeveg i Ben Hesper su uveli ovaj termin 1978. sa značenjem „studiranje informacionih procesa u biološkim sistemima“.[464][465] Ova definicija stavlja bioinformatiku kao polje paralelno sa biofizikom ili biohemijom.[465] Međutim, njena primarna upotreba od kasnih 1980-ih je bilo opisivanje primene informatike i informacionih nauka u analizi bioloških podataka, posebno u oblastima genomike koje se bave DNK sekvenciranjem velikih razmera.
Tokom zadnjih nekoliko dekada brz razvoj genomike i drugih molekularnih istraživačkih tehnologija, u kombinaciji sa razvojem informacionih tehnologija su proizveli ogromne količine molekularno bioloških informacija. Razvoj tehnika za skladištenje i pretraživanje DNK sekvenci su doveli do široke primene sofisticirane informatike, posebno algoritama za pretragu nizova, mašinskog učenja i teorije baza podataka.[466] Pretraživanje nizova ili primena algoritama podudaranja, koji nalaze pojavu sekvence slova unutar veće sekvence slova, su razvijeni specifično za pretraživanje nukleotidnih sekvenci.[467] DNK sekvenca se može poravnati sa drugim DNK sekvencama da bi se identifikovale homologne sekvence i locirale specifične mutacije koje ih čine osobenim. Te tehnike, a posebno poravnavanje višestrukih sekvenci, se koriste u studiranju filogenetičkih odnosa i proteinske funkcije.[468]

Podatke koji obuhvataju celokupne genome, kao što su na primer podaci koje je proizveo Projekat ljudskog genoma, je teško koristiti bez zapisa koji identifikuju lokacije gena i regulatornih elemenata na svakom hromozomu. Regioni DNK sekvence koji imaju karakteristične obrasce asocirane sa proteinskim ili RNK kodirajućim genima se mogu identifikovati primenom algoritama za predviđanje gena, koji omogućavaju formiranje hipoteza o postojanju specifičnih genskih proizvoda i o njihovim mogućim funkcijama u pojedinom organizmu pre nego što dođe do njihove eksperimentalne identifikacije i izolacije.[469] Poređenja celokupnih genoma mogu da daju indikacije o evolucionoj istoriji pojedinog organizma i omoguće razmatranje kompleksnih evolucionih događaja.
Bioinformatika je primenjivana od samog početka „genomske revolucije“. Ona se koristi u formiranju i održavanju baza podataka za skladištenje bioloških informacija, kao što su nukleotidne i aminokiselinske sekvence. Razvoj tog tipa baza podataka se sastoji ne samo od projektovanja strukture podataka, nego i od razvoja kompleksnih interfejsa koji omogućavaju pristup postojećim podacima, kao i unosa novih ili revidiranih podataka.
Da bi se izučavalo kako se normalne ćelijske aktivnosti menjaju u različitim stanjima bolesti, biološki podaci se moraju kombinovati. Na taj način se formira sveobuhvatna slika tih aktivnosti. Polje bioinformatike je evoluiralo tako što je fokus stavljen na analizu i interpretaciju mnoštva različitih tipova podataka. To obuhvata nukleotidne i aminokiselinske sekvence, proteinske domene, i proteinske strukture.[470] Sam proces analiziranja i interpretacije podataka se naziva računska biologija. Važne poddiscipline u okviru bioinformatike i računske biologije su:
- razvoj i implementacija oruđa koja omogućavaju efikasan pristup, korišćenje i upravljanje različitim tipovima informacija.
- razvoj novih algoritama i statističkih tehnika za određivanje odnosa i relacija među članovima velikih grupa podataka. Na primer, metodi za lociranje gena u sekvencama, predviđanje proteinske strukture i/ili funkcije, i grupisanje proteinskih sekvenci u familije srodnih sekvenci.
Primarni cilj bioinformatike je povećavanje razumevanja bioloških procesa. Ono što je izdvaja od drugih pristupa je njen fokus na razvoju i primeni računarski intenzivnih tehnika za postizanje ovog cilja. Neki od primera toga su: algoritmi za prepoznavanje obrazaca, analizu podataka i mašinsko učenje, kao i tehnike za vizuelizaciju bioloških podataka. Glavni istraživački napori u oblasti uključuju poravnavanje sekvenci, nalaženje gena, izučavanje strukture genoma, dizajn lekova, otkrivanje lekova, strukturno poravnavanje proteina, predviđanje proteinske strukture, predviđanje ekspresije gena i protein-protein interakcija, izučavanja genomskih asocijacija i modelovanje evolucije.
Postoje dva fundamentalna načina modelovanja bioloških sistema (npr. živih ćelija).
Strukturna bioinformatika je široka potkategorija bioinformatike. Ona se bavi analizom i predviđanjem trodimenzionalne strukture bioloških makromolekula kao što su proteini, RNK i DNK. Ona proizvodi generalizacije makromolekularnih 3D struktura kao što su poređenja sveukupnih savijanja i lokalnih motiva, principi molekularnog savijanja, evolucije i interakcije vezivanja, i odnosi strukture i funkcije. Ona radi sa eksperimentalno uređenim strukturama i formira računarske modele. Termin strukturna ima ekvivalentno značenje kao i u strukturnoj biologiji. Strukturna bioinformatika se smatra delom računarske strukturne biologije.
DNK nanotehnologija

DNK nanotehnologija koristi jedinstvena svojstva molekulskog prepoznavanja DNK i drugih nukleinskih kiselina da kreira samostalno formirajuće razgranate DNK komplekse koji imaju niz korisnih osobina.[472] Na ovaj način se DNK upotrebljava kao strukturni materijal umesto kao nosilac bioloških informacija. To dovodi do kreiranja dvodimenzionih periodičnih rešetki (u obliku pločica i koristeći „DNK origami“ metod), kao i trodimenzione strukture u obliku poliedra.[473]
Nanomehanički uređaji i algoritamsko samostalno formiranje su isto tako bili demonstrirani.[474] Te DNK strukture su bile korišćene kao templet za organizovanje drugih molekula kao što su nanočestice zlata i streptavidinski proteini.[475]
Konceptualnu osnovu DNK nanotehnologije je položio Nadrijan Siman tokom ranih 1980-ih, a polje je počelo da privlači široko interesovanje početkom i sredinom 2000-tih. Ova oblast počinje da se koristi kao izvor oruđa za rešavanje problema bazne nauke u strukturnoj biologiji i biofizici, uključujući primenu u kristalografiji i spektroskopiji za demonstraciju proteinske strukture. Potencijalne praktične primene u elektronici molekularnih srazmera i nanomedicini se takođe istražuju.
DNK nanotehnologija se ponekad deli u dva preklapajuća potpolja: strukturnu DNK nanotehnologija i dinamičku DNK nanotehnologija. Strukturna DNK nanotehnologija ima fokus na sintezi i karakterizaciji nukleinsko kiselinskih kompleksa i materijala koji se sklapaju u statičko, ravnotežno krajnje stanje. S druge strane, dinamička DNK nanotehnologija se usredsređuje na komplekse sa korisnim neravnotežnim ponašanjem, kao što je sposobnost da se promeni konfiguracije nakon hemijskih i fizičkih stimulusa. Neki kompleksi kombinuju svojstva oba, strukturnog i dinamičkog potpolja, npr. nukleinsko kiselinski nanomehanički uređaji.[476][477]

Kompleksi konstruisani primenom strukturne DNK nanotehnologije koriste razgranate strukture nukleinske kiseline koja sadrži tačke spajanja, za razliku od većine biološke DNK, koja se javlja kao nerazgranati dvostruki heliks. Jedna od najjednostavnijih razgranatih struktura, i prvo napravljena, je četvorostruka raskrsnica koja se sastoji od četiri individualna DNK lanca, čije porcije su komplementarne i imaju specifičan obrazac. Za razliku od prirodnog Holidejovog spoja, svaka ruka u veštačkom nepokretnom spoju ima različitu baznu sekvencu, te je raskrsnica fiksna u određenoj poziciji. Višestruki spojevi se mogu kombinovati u istom molekulu. U najširoj upotrebi su motivi „dvostrukih skretnica“ (DX). Taj motiv se sastoji od dva paralelna dvostruka heliksna domena, gde individualni lanci prelaze iz jednog domena u drugi u dvema tačkama račvanja. Svaka tačka račvanja je sa topološkog gledišta raskrsnica sa četiri ruke, koja je ograničena u jednoj orijentaciji, što je u kontrastu sa fleksibilnim raskrsnicama. Ta krutost čini DX motiv podesnim strukturnim gradivnim blokom za veće DNK komplekse.[479][480]
Dinamička DNK nanotehnologija često koristi mehanizam polaznom tačkom posredovanog zamenjivanja lanca da bi se omogućila rekonfiguracija kompleksa nukleinske kiseline. U ovoj reakciji, lanac se vezuje za region jednolančane početne tačke dvolančanog kompleksa, i zatim zamenjuje jedan od lanaca vezanih za originalni kompleks putem procesa migracije grane. Sveukupni efekat je zamena jednog lanca kompleksa, što omogućava da prisustvo prvog lanca deluje kao prekidač za kontrolu rekonfiguracije kompleksa.[476] Moguće je napraviti strukture i uređaje koristeći funkcionalne nukleinske kiseline kao što su dezoksiribozimi i ribozimi, koji mogu da izvode hemijske reakcije, i aptamere, koji mogu da vežu specifične proteine ili male molekule.[481]
Alternativni genetički polimeri
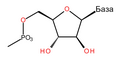 ANK (arabinoza) |
 FANK (2´-fluoroarabinoza) |
 TNK (treoza) |
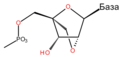 LNK („zaključana“ riboza) |
 CeNK (cikloheksen) |
 HNK (anhidroheksitol) |
Skladištenje i manipulacija genetičke informacije se u prirodi oslanjaju na samo dva polimera, DNK i RNK. Nije u potpunosti razjašnjeno da li je ova njihova uloga odražava evolucionu istoriju ili fundamentalna funkcionalna ograničenja molekula. Primenom projektovanih polimeraza se može pokazati da se genetička informacija može čuvati i koristiti primenom niza alternativnih genetičkih polimera baziranih na jednostavnoj arhitekturi nukleinskih kiselina koje se ne javljaju u prirodi, ksenonukleinskih kiselina (KNK). Podesnim izborom KNK aptamera, koji se vezuju za svoje ciljeve sa visokim afinitetom i specifičnošću, pokazano je da pored naslednosti, specifične ksenonukleinske kiseline imaju sposobnost Darvinske evolucije i formiranja definisanih struktura. Nedavno je objavljena studija u kojoj su opisana svojstva šest alternativnih genetičkih polimera koji se mogu koristiti za čuvanje i propagiranje informacije. Iz toga proizilazi da naslednost i evolucija, dva obeležja života, nisu ograničena na DNK i RNK nego da su verovatno pojavni oblici polimera koji imaju sposobnost skladištenja informacija.[482]
Razvoj ksenonukleinskih kiselina označava početak ere sintetičke genetike, sa implikacijama za astrobiologiju, biotehnologiju i razumevanje života. Dosadašnji koraci izvan biološke genetike su skromni. Oni obuhvataju KNK molekule, koji su analogni sa biološkim nukleinskim kiselinama. Njihov šećer ili šećeru slična komponenta nije riboza. Ona je zamenjena različitim šećerima sa pet ugljenika (arabinozom u ANK, 2´-fluoroarabinozom u FANK), šećerom sa četiri ugljenika (treozom u TNK), i „zaključanim“ riboznim analogom u LNK, ili strukturama sa šestočlanim prstenom (cikloheksenom u CeNK, anhidroheksitolom u HNK). Izučavanje ksenonukleinskih kiselina je inspirisano pitanjem o prvom genetičkom polimeru na Zemlji. Možda je to bila RNK, ali je isto tako moguće da je to bila neka od jednostavnijih struktura koje bi bile dostupnije putem prebiotičke sinteze.[483] TNK i glikolna nukleinska kiselina (GNK) su primarni kandidati.[484][485] Drugi razlog za izučavanje ksenonukleinskih kiselina je njihova primena u obliku antisense agenasa koji se vezuju i inhibiraju funkcije bioloških ribonukleinskih kiselina. Svih šest Pinheirovih ksenonukleinskih kiselina se vezuju za komplementarne RNK i DNK, i otporne su na degradaciju biološkim nukleazama.[482] Konstrukcija genetičkog sistema baziranog na alternativnim hemijskim platformama mogla bi konačno da dovede do sinteze novih formi života.
Sadašnji stepen razvoja omogućava replikaciju KNK reverznim transkribovanjem do DNK, njenim umnožavanjem putem PCR, i zatim transkribovanjem DNK nazad u KNK. U svakom stepenu se koriste polimeraze. Konverzija u DNK je neophodna za amplifikaciju.[486] Ključni korak u XNK istraživanjima je bio razvoj varijanti polimeraza koji imaju sposobnost kopiranja informacije između XNK i DNK. XNK polimeri sa više od 70 podjedinica i skoro svaka sekvenca se može kopirati, sa prosečnom tačnošću od 95% (za LNK) do 99,6 % (za CeNK). Taj profil je dovoljan za sprovođenje usmerene evolucije funkcionalnih KNK molekula. Primenom tog procesa na HNK polimere je pokazano je da oni mogu da evoluiraju u laboratoriji tako da se dobijaju funkcionalni molekuli (aptameri) koji se čvrsto i specifično vezuju za željeni RNK ili proteinski molekul. Razvoj polimeraza koje mogu da kopiraju KNK na njen sopstveni komplement, ili da kopiraju informacije između dve različite KNK, je u toku. Do sada, FANK i CeNK su bile kopirane na njihove komplemente, i CeNK je bila kopirana na HNK, ali su ti procesi znatno manje efikasni nego kopiranje između KNK i DNK.
Još jedan važan razlog za nastavljanje razvoja funkcionalnih KNK je dobijanje jedinjenja za potencijalnu primenu u nauci o materijalima, molekularnoj dijagnostici, i lekovima. Aptameri nukleinskih kiselina su već našli široku primenu u tim oblastima. Međutim, RNK i DNK su podložni dejstvu bioloških nukleaza, te se oni moraju modifikovati da bi se održali u prirodnim uslovima. KNK molekuli su veštački te nisu podložni dejstvu nukleaza. Korist od njihovih neobičnih hemijskih osobina se mora odmeravati sa njihovom većom cenom, neposredno, i u kontekstu rada u neistraženom domenu KNK biohemije.
Populaciona genetika
DNK tokom vremena sakupi mutacije koje se prenose sa kolena na koleno, te stoga DNK sadrži istorijske informacije. Poređenjem DNK sekvenci se može pratiti evoluciona istorija organizama, njihova filogenija.[487] Ovo područje filogenetike je bogat izvor informacija o evolucionoj biologiji. Populaciono genetički uvid u istoriju pojedinih populacija se može dobiti putem poređenja DNK sekvenci unutar vrsta. Taj pristup nalazi primenu u širokom spektru studija od ekološke genetike do antropologije. Populaciona genetika je studija distribucije frekvencije alela i promena pod uticajem četiri glavna evoluciona procesa: prirodna selekcija, genetički drift, mutacija i protok gena. Ona isto tako uzima u obzir faktore rekombinacije, populacione potpodele i populacione strukture. Populaciona genetika pokušava da objasni fenomene poput adaptacije i specijacije.
Genetička genealogija
Genetička genealogija je primena genetike u tradicionalnoj genealogiji. Genetička genealogija obuhvata upotrebu genealoškog DNK testiranja radi određivanja nivoa genetičkih odnosa između pojedinaca. Dva najčešće korišćena tipa genetičkih genealoških testova su Y-DNK (očinska linija)[488] i mtDNK[489] (materinska linija).[488]
Ovi testovi se sastoje od poređenja pojedinih DNK sekvenci para osoba da bi se procenila verovatnoća da oni imaju zajedničkog prethodnika u genealoškom vremenskom okviru. Primenom Bajesovog modela koji je objavio Brus Volš, za procenu broja generacija koje razdvajaju dve osobe od njihovog najskorijeg zajedničkog pretka.[490]
Y-DNK testiranje obuhvata kratka tandemna ponavljanja (STR), a ponekad i testiranje jednonuklearnih polimorfizama (SNP) Y-hromozoma. Ovaj hromozom je prisutan samo kod muškaraca, te daje informacije samo o očinskoj liniji. Ovi testovi mogu da pruže uvid u nedavno (STR) i drevno (SNP) genetičko poreklo. Y-hromozomni STR test sadrži haplotip koji je sličan kod svih muških potomaka zajedničkog muškog pretka. SNP testovi se koriste za svrstavanje ljudi u očinske haplogrupe, koje definišu znatno veću genetičku populaciju.
mtDNK testiranje obuhvata sekvenciranje HVR-1 regiona, HVR-2 regiona ili oba. mtDNK test može takođe da sadrži dodatne SNP-ove koji su potrebni za svrstavanje ljudi u materinske haplogrupe, ili čak kompletnu mtDNK.
Rezultati Y-DNK ili mtDNK testova se mogu porediti sa rezultatima drugih osoba upotrebom privatnih ili javnih DNK baza podataka.
Istorija DNK istraživanja
DNK je prvi izolovao švajcarski lekar Fridrih Mišer, koji je 1869. otkrio mikroskopsku supstancu u gnoju odbačenih zavoja. Ona se nalazila u nukleusima ćelija, te ju je on nazvao „nuklein“.[491] Albreht Kosel je 1878. izolovao neproteinsku komponentu „nukleina“, nukleinsku kiselinu. On je kasnije izolovao i pet primarnih nukleobaza.[492] Fibus Lavin je 1919. identifikovao bazu, šećer i fosfat nukleotidne jedinice.[493] Lavin je došao do zaključka da se DNK sastoji od niza nukleotidnih jedinica povezanih fosfatnim grupama. Međutim, on je smatrao da je lanac kratak i da se baze ponavljaju u fiksnom poretku. Vilijam Astburi je 1937. proizveo prve Rendgenske difrakcione obrasce koji su pokazali da DNK ima uređenu strukturu.[494][495]
Nikolaj Koltsov je 1927. predložio da se nasledne osobine mogu prenositi putem „gigantskog molekula za nasleđivanje“ koji bi se sastojao od „dva komplementarna lanca koji be se kopirali koristeći jedan lanac kao templet“.[496] Frederik Grifit je 1928. otkrio da se svojstvo „glatkog“ oblika Pneumococcus bakterije može preneti na bakterije „neravnog“ oblika iste vrste putem mešanja mrtvih glatkih bakterija sa živim grubim bakterijama.[497] Taj sistem je pružio prvu jasnu indikaciju da DNK nosi genetičke informacije. Osvald Ejveri zajedno sa saradnicima Kolinom Maklaudom i Maklinom Mekartijem su identifikovali DNK kao nosioca transformirajućeg principa 1943.[498] Uloga DNK molekula u nasleđivanju je potvrđena 1952, kad su Alfred Herši i Marta Čejs pokazali da je DNK genetički materijal T2 faga.[499]
Tokom 1950-ih tri grupe su radile na određivanju strukture DNK. Sa radom je prvo započela grupa sa Kings koledža u Londonu koju je predvodio Moris Vilkins i kojoj se kasnije pridružila Rozalind Franklin. Druga grupa se sastojala of Fransisa Krika i Džejmsa Votsona u Kembridžu. Treća grupa je radila na Kaltehu i bila je predvođena Linusom Paulingom. Krik i Votson su izradili fizičke modele koristeći metalne poluge i kugle, u kojima se inkorporisali poznate hemijske strukture nukleotida, kao i poznate pozicije veza koje spajaju nukleotide duž polimera. Na Kings koledžu Moris Vilkins i Rosalind Franklin su izučavali rendgenske difrakcione obrasce DNK niti. Od tri grupe, jedino je londonska grupa bila u mogućnosti da proizvede difrakcione obrasce zadovoljavajućeg kvaliteta i da tako generiše dovoljnu količinu kvantitativnih podataka o strukturi.
Džejms D. Votson i Fransis Krik su 1953. predložili prvi korektan model dvostrukog heliksa DNK strukture.[12] Njihov model je bio baziran na samo jednom rendgenskom difrakcionom snimku (obeleženom kao „Fotografija 51“)[500] koji su snimili Rosalind Franklin i Rejmond Gosling maja 1952, kao i informaciji da su DNK baze sparene, što je proisteklo iz privatne komunikacije sa Ervinom Čargafom tokom prethodnih godina. Čargafova pravila su imala veoma važnu ulogu u određivanju konfiguracije dvostrukog heliksa za B-DNK kao i za A-DNK.
Eksperimentalni dokazi koji podržavaju model Votsona i Krika su objavljeni u seriji od pet članaka u istom izdanju časopisa Priroda.[501] Među njima Franklinov i Goslingov članak su bili prva publikacija njihovih Rendgenskih difrakcionih podataka i originalni metod analize koji je delom podržavao model Votson i Krika.[85][502] To izdanje je takođe sadržalo članak o DNK strukturi Morisa Vilkinsa i dvoje njegovih saradnika, čija analiza in vivo B-DNK Rendgenskih obrazaca je takođe podržavala prisustvo in vivo dvostrukog heliksa u DNK konfiguracije, kao što su predložili Krik i Votson.[86] Franklin je umrla 1958. Votson, Krik i Vilkins su dobili Nobelovu nagradu za fiziologiju ili medicinu 1962.[503][504]
U jednoj uticajnoj prezentaciji iz 1957, Krik je izložio centralnu dogmu molekularne biologije, koja je predskazala odnos između DNK, RNK i proteina, i artikulisala hipotezu adaptera.[505] Finalna potvrda replikacionog mehanizma koji je proizašao iz strukture dvostrukog heliksa je usledila 1958. u obliku Meselson-Stahlovog eksperimenta.[506] Daljim radom Krika i njegovih saradnika je pokazano da je genetički kod baziran na nepreklapajućim tripletima baza, koji se nazivaju kodoni. Genetički kod su dešifrovali Har Gobind Korana, Robert Holi i Maršal Varen Nirenberg.[507] Ovi nalazi označavaju rođenje polja molekularne biologije.
Vidi još
Reference
Literatura
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Dumanović, J, marinković, D, Denić, M: Genetički rečnik, Beograd, 1985.
- Kosanović, M, Diklić, V: Odabrana poglavlja iz humane genetike, Beograd, 1986.
- Lazarević, M: Ogledi iz medicinske genetike, Beograd, 1986.
- Marinković, D, Tucić, N, Kekić, V: Genetika, Naučna knjiga, Beograd
- Matić, Gordana: Osnovi molekularne biologije, Zavet, Beograd, 1997.
- Ridli, M: Genom — autobiografija vrste u 23 poglavlja, Plato, Beograd, 2001.
- Tatić, S, Kostić, G, Tatić, B: Humani genom, ZUNS, Beograd, 2002.
- Tucić, N, Matić, Gordana: O genima i ljudima, Centar za primenjenu psihologiju, Beograd, 2002.
- Šerban, Nada: ćelija — strukture i oblici, ZUNS, Beograd, 2001
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Horace Freeland Judson 1979. The Eighth Day of Creation: Makers of the Revolution in Biology. Touchstone Books, Šablon:ISBN. 2nd edition: Cold Spring Harbor Laboratory Press, 1996 paperback: Šablon:ISBN.
- Šablon:Cite book, first published in October 1974 by MacMillan, with foreword by Francis Crick;the definitive DNA textbook,revised in 1994 with a 9 page postscript
- Micklas, David. 2003. DNA Science: A First Course. Cold Spring Harbor Press:. Šablon:ISBN.
- Šablon:Cite book
- Šablon:Cite book
- Rosenfeld, Israel. 2010. DNA: A Graphic Guide to the Molecule that Shook the World. Columbia University Press:. Šablon:ISBN.
- Schultz, Mark and Zander Cannon. 2009. The Stuff of Life: A Graphic Guide to Genetics and DNA. Hill and Wang: Šablon:ISBN
- Šablon:Cite book
- James D. Watson 2004. DNA: The Secret of Life. Random House:. Šablon:ISBN.
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book
- Šablon:Cite book